Menschliche genetische Variation - Human genetic variation
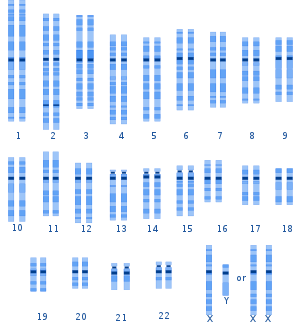
Die genetische Variation des Menschen sind die genetischen Unterschiede in und zwischen Populationen . Es kann mehrere Varianten eines beliebigen Gens in der menschlichen Population ( Allele ) geben, eine Situation, die als Polymorphismus bezeichnet wird .
Keine zwei Menschen sind genetisch identisch. Selbst eineiige Zwillinge (die sich aus einer Zygote entwickeln) weisen aufgrund von während der Entwicklung auftretenden Mutationen und Variationen der Genkopienzahl selten genetische Unterschiede auf . Unterschiede zwischen Individuen, sogar eng verwandten Individuen, sind der Schlüssel zu Techniken wie dem genetischen Fingerabdruck . Mit Stand 2017 sind insgesamt 324 Millionen Varianten aus sequenzierten menschlichen Genomen bekannt . Im Jahr 2015 wurde der typische Unterschied zwischen dem Genom eines Individuums und dem Referenzgenom auf 20 Millionen Basenpaare (oder 0,6% der insgesamt 3,2 Milliarden Basenpaare) geschätzt.
Allele treten in verschiedenen menschlichen Populationen mit unterschiedlicher Häufigkeit auf. Populationen, die geografisch und von vornherein weiter entfernt liegen , unterscheiden sich tendenziell stärker. Die Unterschiede zwischen den Populationen machen einen kleinen Teil der gesamten genetischen Variation des Menschen aus. Populationen unterscheiden sich auch in der Menge der Variation zwischen ihren Mitgliedern. Die größte Divergenz zwischen den Populationen findet sich in Subsahara-Afrika , was mit der jüngsten afrikanischen Herkunft nichtafrikanischer Populationen übereinstimmt . Die Populationen unterscheiden sich auch in dem Anteil und dem Ort der introgressierten Gene, die sie durch archaische Beimischung sowohl innerhalb als auch außerhalb Afrikas erhalten haben.
Das Studium der genetischen Variation beim Menschen hat evolutionäre Bedeutung und medizinische Anwendungen. Es kann Wissenschaftlern helfen, die Migrationen der alten menschlichen Bevölkerung zu verstehen und zu verstehen, wie menschliche Gruppen biologisch miteinander verwandt sind. Für die Medizin kann die Untersuchung der genetischen Variation beim Menschen wichtig sein, da einige krankheitsverursachende Allele häufiger bei Menschen aus bestimmten geografischen Regionen auftreten. Neue Erkenntnisse zeigen, dass jeder Mensch im Vergleich zu seinen Eltern durchschnittlich 60 neue Mutationen aufweist.
Variationsursachen
Ursachen für Unterschiede zwischen Individuen sind unabhängige Sortierung , der Austausch von Genen (Cross-Over und Rekombination) während der Reproduktion (durch Meiose ) und verschiedene Mutationsereignisse .
Es gibt mindestens drei Gründe, warum genetische Variation zwischen Populationen existiert. Natürliche Selektion kann Individuen in einer bestimmten Umgebung einen adaptiven Vorteil verleihen, wenn ein Allel einen Wettbewerbsvorteil bietet. Allele, die ausgewählt werden, treten wahrscheinlich nur in den geographischen Regionen auf, in denen sie einen Vorteil verleihen. Ein zweiter wichtiger Prozess ist die genetische Drift , die die Wirkung zufälliger Veränderungen im Genpool ist, unter Bedingungen, bei denen die meisten Mutationen neutral sind (dh sie scheinen keine positive oder negative selektive Wirkung auf den Organismus zu haben). Schließlich weisen kleine Migrantenpopulationen statistische Unterschiede – den sogenannten Gründereffekt – von der Gesamtbevölkerung auf, aus der sie stammen; Wenn diese Migranten neue Gebiete besiedeln, unterscheidet sich ihre Nachkommenpopulation typischerweise von ihrer Herkunftspopulation: unterschiedliche Gene überwiegen und sie ist genetisch weniger vielfältig.
Beim Menschen ist die Hauptursache genetische Drift . Serielle Gründereffekte und eine frühere kleine Populationsgröße (die die Wahrscheinlichkeit einer genetischen Drift erhöht) haben möglicherweise einen wichtigen Einfluss auf die neutralen Unterschiede zwischen den Populationen. Die zweite Hauptursache für genetische Variation liegt in der hohen Neutralität der meisten Mutationen . Eine kleine, aber bedeutende Anzahl von Genen scheint kürzlich einer natürlichen Selektion unterzogen worden zu sein, und dieser Selektionsdruck ist manchmal spezifisch für eine Region.
Variationsmaße
Genetische Variation beim Menschen tritt auf vielen Ebenen auf, von groben Veränderungen des menschlichen Karyotyps bis hin zu einzelnen Nukleotidänderungen . Chromosomenanomalien werden bei 1 von 160 Lebendgeburten beim Menschen festgestellt. Abgesehen von Störungen der Geschlechtschromosomen führen die meisten Fälle von Aneuploidie zum Tod des sich entwickelnden Fötus ( Fehlgeburt ); die häufigsten extra autosomalen Chromosomen bei Lebendgeburten sind 21 , 18 und 13 .
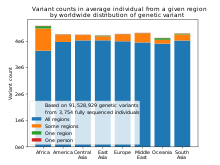
Nukleotiddiversität ist der durchschnittliche Anteil von Nukleotiden, die sich zwischen zwei Individuen unterscheiden. Ab 2004 wurde die menschliche Nukleotiddiversität auf 0,1% bis 0,4% der Basenpaare geschätzt . Im Jahr 2015 stellte das 1000 Genomes Project , das tausend Individuen aus 26 menschlichen Populationen sequenzierte, fest, dass „ein typisches [individuelles] Genom sich von dem menschlichen Referenzgenom an 4,1 Millionen bis 5,0 Millionen Stellen unterscheidet … mit Auswirkungen auf 20 Millionen Sequenzbasen“; letztere Zahl entspricht 0,6% der Gesamtzahl der Basenpaare. Fast alle (>99,9%) dieser Stellen sind kleine Unterschiede, entweder einzelne Nukleotidpolymorphismen oder kurze Insertionen oder Deletionen ( Indels ) in der genetischen Sequenz, aber strukturelle Variationen machen eine größere Anzahl von Basenpaaren aus als die SNPs und Indels.
Ab 2017 listete die Single Nucleotide Polymorphism Database ( dbSNP ), die SNP und andere Varianten auflistet, 324 Millionen Varianten auf, die in sequenzierten menschlichen Genomen gefunden wurden.
Einzelnukleotidpolymorphismen
Ein einzelner Nukleotidpolymorphismus (SNP) ist ein Unterschied in einem einzelnen Nukleotid zwischen Mitgliedern einer Spezies, der bei mindestens 1% der Population auftritt. Die 2.504 Individuen, die durch das 1000 Genomes Project charakterisiert wurden, hatten 84,7 Millionen SNPs darunter. SNPs sind die häufigste Art von Sequenzvariation, die 1998 auf 90% aller Sequenzvarianten geschätzt wurde. Andere Sequenzvariationen sind Einzelbasenaustausche, Deletionen und Insertionen. SNPs treten im Durchschnitt etwa alle 100 bis 300 Basen auf und sind somit die Hauptquelle der Heterogenität.
Ein funktioneller oder nicht-synonymer SNP ist einer, der einen Faktor wie das Spleißen von Genen oder Boten-RNA beeinflusst und so einen phänotypischen Unterschied zwischen den Mitgliedern der Spezies verursacht. Etwa 3% bis 5% der menschlichen SNPs sind funktionell (siehe International HapMap Project ). Neutrale oder synonyme SNPs sind aufgrund ihrer schieren Zahl und der stabilen Vererbung über Generationen hinweg noch immer als genetische Marker in genomweiten Assoziationsstudien nützlich .
Ein kodierender SNP tritt innerhalb eines Gens auf. Es gibt 105 Human Reference SNPs, die zu vorzeitigen Stoppcodons in 103 Genen führen. Dies entspricht 0,5% der kodierenden SNPs. Sie entstehen durch segmentale Duplikation im Genom. Diese SNPs führen zu einem Proteinverlust, jedoch sind alle diese SNP-Allele üblich und werden bei negativer Selektion nicht gereinigt .
Strukturelle Variation
Strukturelle Variation ist die Variation in der Struktur des Chromosoms eines Organismus . Strukturelle Variationen, wie Kopienzahlvariationen und Deletionen , Inversionen , Insertionen und Duplikationen , sind für viel mehr menschliche genetische Variation verantwortlich als die Diversität einzelner Nukleotide. Dies wurde 2007 aus der Analyse der diploiden vollständigen Sequenzen der Genome zweier Menschen geschlossen: Craig Venter und James D. Watson . Dies fügte den beiden haploiden Sequenzen hinzu, die Verschmelzungen von Sequenzen vieler Individuen waren, die vom Human Genome Project bzw. Celera Genomics veröffentlicht wurden.
Laut dem 1000 Genomes Project hat ein typischer Mensch 2.100 bis 2.500 strukturelle Variationen, die ungefähr 1.000 große Deletionen, 160 Kopienzahlvarianten, 915 Alu- Insertionen, 128 L1- Insertionen, 51 SVA-Insertionen, 4 NUMTs und 10 Inversionen umfassen.
Nummernvariation kopieren
Eine Kopienzahlvariation (CNV) ist ein Unterschied im Genom aufgrund des Löschens oder Duplizierens großer DNA-Regionen auf einem Chromosom. Es wird geschätzt, dass sich 0,4% der Genome von nicht verwandten Menschen hinsichtlich der Kopienzahl unterscheiden. Wenn die Variation der Kopienzahl berücksichtigt wird, wird die genetische Variation von Mensch zu Mensch auf mindestens 0,5 % geschätzt (99,5% Ähnlichkeit). Variationen der Kopienzahl werden vererbt, können aber auch während der Entwicklung auftreten.
Eine visuelle Karte mit den Regionen mit hoher genomischer Variation der modernen menschlichen Referenzanordnung relativ zu einem Neandertaler von 50.000 wurde von Pratas et al.
Epigenetik
Epigenetische Variation ist eine Variation der chemischen Markierungen, die an DNA anhaften und beeinflussen, wie Gene gelesen werden. Die Tags, "epigenetische Markierungen genannt, fungieren als Schalter, die steuern, wie Gene gelesen werden können." Bei einigen Allelen können der epigenetische Zustand der DNA und der zugehörige Phänotyp über Generationen von Individuen vererbt werden .
Genetische Variabilität
Die genetische Variabilität ist ein Maß für die Tendenz einzelner Genotypen in einer Population, sich voneinander zu unterscheiden. Variabilität unterscheidet sich von genetischer Diversität , d. h. der Menge an Variation, die in einer bestimmten Population beobachtet wird. Die Variabilität eines Merkmals gibt an, wie stark dieses Merkmal als Reaktion auf Umwelt- und genetische Einflüsse tendenziell variiert .
Clines
In der Biologie ist eine Kline ein Kontinuum von Arten , Populationen, Varietäten oder Formen von Organismen, die allmähliche phänotypische und/oder genetische Unterschiede in einem geografischen Gebiet aufweisen, typischerweise als Folge von Umweltheterogenität. Bei der wissenschaftlichen Erforschung der genetischen Variation beim Menschen kann eine Gen-Kline genau definiert und quantitativen Metriken unterzogen werden.
Haplogruppen
In der Untersuchung der molekularen Evolution ist eine Haplogruppe eine Gruppe ähnlicher Haplotypen , die einen gemeinsamen Vorfahren mit einer Single Nucleotid Polymorphism (SNP)-Mutation teilen . Die Untersuchung von Haplogruppen liefert Informationen über die Ursprünge der Vorfahren, die Tausende von Jahren zurückreichen.
Die am häufigsten untersuchten menschlichen Haplogruppen sind Y-Chromosom (Y-DNA) Haplogruppen und mitochondriale DNA (mtDNA) Haplogruppen , die beide verwendet werden können, um genetische Populationen zu definieren. Y-DNA wird ausschließlich entlang der patrilinearen Linie vom Vater an den Sohn weitergegeben, während mtDNA entlang der matrilinearen Linie von der Mutter an die Tochter oder den Sohn weitergegeben wird. Die Y-DNA und mtDNA können sich bei jeder Generation durch zufällige Mutation ändern.
Tandemwiederholungen mit variabler Anzahl
Eine Tandemwiederholung mit variabler Anzahl (VNTR) ist die Variation der Länge einer Tandemwiederholung . Ein Tandem-Repeat ist die benachbarte Wiederholung einer kurzen Nukleotidsequenz . Tandem-Wiederholungen existieren auf vielen Chromosomen , und ihre Länge variiert von Person zu Person. Jede Variante fungiert als ein vererbtes Allel , so dass sie zur persönlichen oder elterlichen Identifizierung verwendet werden. Ihre Analyse ist nützlich in der genetischen und biologischen Forschung, in der Forensik und beim DNA-Fingerabdruck .
Kurze Tandemwiederholungen (etwa 5 Basenpaare) werden Mikrosatelliten genannt , während längere Minisatelliten genannt werden .
Geschichte und geografische Verteilung
Neuer afrikanischer Ursprung des modernen Menschen
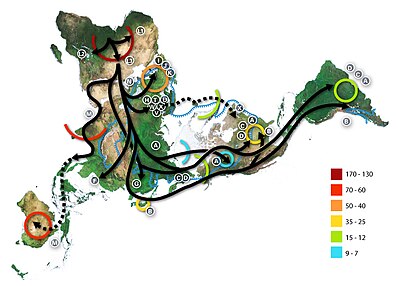
Das Paradigma des jüngsten afrikanischen Ursprungs des modernen Menschen geht davon aus, dass sich die nichtafrikanischen Populationen des anatomisch modernen Menschen nach 70.000 Jahren zerstreut haben. Die Ausbreitung innerhalb Afrikas erfolgte deutlich früher, vor mindestens 130.000 Jahren. Die „out of Africa“ -Theorie stammt aus dem 19. Jahrhundert, als vorläufigen Vorschlag in Charles Darwins Descent of Man , blieb aber spekulativ , bis die 1980er Jahre , als es durch das Studium des heutigen Mitochondrien - DNA, kombiniert mit dem Nachweis von unterstützt physikalischer Anthropologie von archaischen Exemplaren .
Laut einer Studie über die Variation der Y-Chromosomensequenz aus dem Jahr 2000 gehen menschliche Y-Chromosomen auf Afrika zurück, und die Nachkommen der abgeleiteten Linie verließen Afrika und wurden schließlich in Eurasien durch archaische menschliche Y-Chromosomen ersetzt. Die Studie zeigt auch, dass eine Minderheit der heutigen Bevölkerungen in Ostafrika und den Khoisan die Nachkommen der ältesten Patrilinien der anatomisch modernen Menschen sind, die Afrika vor 35.000 bis 89.000 Jahren verlassen haben. Andere Beweise, die die Theorie stützen, sind, dass die Variationen der Schädelmaße mit der Entfernung von Afrika im gleichen Maße abnehmen wie die genetische Vielfalt. Die genetische Vielfalt des Menschen nimmt in einheimischen Populationen mit Migrationsentfernung von Afrika ab, und es wird angenommen, dass dies auf Engpässe während der menschlichen Migration zurückzuführen ist, bei denen es sich um Ereignisse handelt, die die Populationsgröße vorübergehend verringern.
Eine genetische Clustering-Studie aus dem Jahr 2009, bei der 1327 polymorphe Marker in verschiedenen afrikanischen Populationen genotypisiert wurden, identifizierte sechs Vorfahren-Cluster. Die Clusterung entsprach eng mit Ethnizität, Kultur und Sprache. Eine Studie zur Sequenzierung des gesamten Genoms aus dem Jahr 2018 der Weltbevölkerung beobachtete ähnliche Cluster unter den Populationen in Afrika. Bei K=9 definierten unterschiedliche Ahnenkomponenten die afroasiatisch sprechenden Populationen, die Nordafrika und Nordostafrika bewohnen ; die Nilo-Sahara- sprechende Bevölkerung in Nordostafrika und Ostafrika ; die Ari- Populationen in Nordostafrika; die Niger-Kongo- sprechende Bevölkerung in West-Zentralafrika, Westafrika , Ostafrika und im südlichen Afrika ; die Pygmäen- Populationen in Zentralafrika ; und die Khoisan- Populationen im südlichen Afrika.
Populationsgenetik
Aufgrund der gemeinsamen Abstammung aller Menschen weisen nur wenige Varianten große Häufigkeitsunterschiede zwischen den Populationen auf. Einige seltene Varianten in der Weltbevölkerung sind jedoch in mindestens einer Population viel häufiger (mehr als 5%).
Es wird allgemein angenommen, dass die frühen Menschen Afrika verlassen haben und somit vor ihrer afrikanisch-eurasischen Divergenz vor etwa 100.000 Jahren (ca. 3.000 Generationen) einen Populationsengpass passiert haben müssen. Die schnelle Expansion einer bisher kleinen Population hat zwei wichtige Auswirkungen auf die Verbreitung genetischer Variation. Erstens tritt der sogenannte Gründereffekt auf, wenn Gründerpopulationen nur eine Teilmenge der genetischen Variation ihrer Vorfahrenpopulation mitbringen. Zweitens wird die Wahrscheinlichkeit, dass sich zwei Individuen aus unterschiedlichen Gründerpopulationen paaren, kleiner, wenn die Gründer geografisch stärker voneinander getrennt sind. Die Wirkung dieser assortativen Paarung besteht darin, den Genfluss zwischen geografischen Gruppen zu reduzieren und die genetische Distanz zwischen den Gruppen zu vergrößern.
Die Ausbreitung des Menschen aus Afrika beeinflusste die Verbreitung der genetischen Variation auf zwei andere Weisen. Erstens erfahren kleinere (Gründer-)Populationen eine größere genetische Drift aufgrund erhöhter Schwankungen neutraler Polymorphismen. Zweitens wurden neue Polymorphismen, die in einer Gruppe auftraten, weniger wahrscheinlich auf andere Gruppen übertragen, da der Genfluss eingeschränkt war.
Bevölkerungen in Afrika weisen tendenziell ein geringeres Kopplungsungleichgewicht auf als Bevölkerungen außerhalb Afrikas, teilweise aufgrund der größeren Bevölkerungszahl in Afrika im Laufe der Menschheitsgeschichte und teilweise aufgrund der Anzahl der modernen Menschen, die Afrika verlassen haben, um den Rest von Afrika zu kolonisieren die Welt scheint relativ niedrig gewesen zu sein. Im Gegensatz dazu können Populationen, die in der Vergangenheit eine dramatische Größenreduktion oder schnelle Expansion erfahren haben, und Populationen, die durch die Mischung von zuvor getrennten Vorfahrengruppen gebildet wurden, ungewöhnlich hohe Kopplungsungleichgewichte aufweisen
Verteilung der Variation

Die Verteilung genetischer Varianten innerhalb und zwischen menschlichen Populationen kann aufgrund der Schwierigkeit, eine „Population“ zu definieren, der klinalen Natur der Variation und der Heterogenität über das Genom nicht prägnant beschrieben werden (Long und Kittles 2003). Im Allgemeinen gibt es jedoch durchschnittlich 85 % der genetischen Variation innerhalb lokaler Populationen, ~7 % zwischen lokalen Populationen innerhalb desselben Kontinents und ~8 % der Variation treten zwischen großen Gruppen auf, die auf verschiedenen Kontinenten leben. Die neuere Theorie des afrikanischen Ursprungs für den Menschen würde vorhersagen, dass es in Afrika viel mehr Vielfalt gibt als anderswo und dass die Vielfalt abnehmen sollte, je weiter von Afrika entfernt eine Population untersucht wird.
Phänotypische Variation
Afrika südlich der Sahara weist die größte genetische Vielfalt des Menschen auf, und das gleiche gilt nachweislich für die phänotypische Variation der Schädelform. Der Phänotyp ist durch die Genexpression mit dem Genotyp verbunden . Die genetische Vielfalt nimmt mit der Migrationsentfernung von dieser Region, von der viele Wissenschaftler glauben, dass sie der Ursprung des modernen Menschen ist, sanft ab, und diese Abnahme wird durch eine Abnahme der phänotypischen Variation widergespiegelt. Schädelmessungen sind ein Beispiel für ein physisches Attribut, dessen Variation innerhalb der Bevölkerung mit der Entfernung von Afrika abnimmt.
Die Verteilung vieler physischer Merkmale ähnelt der Verteilung der genetischen Variation innerhalb und zwischen menschlichen Populationen ( American Association of Physical Anthropologists 1996; Keita und Kittles 1997). Zum Beispiel treten ~90% der Variationen der menschlichen Kopfformen innerhalb kontinentaler Gruppen auf und ~10% trennen Gruppen, wobei eine größere Variabilität der Kopfform bei Individuen mit jüngsten afrikanischen Vorfahren besteht (Relethford 2002).
Eine prominente Ausnahme von der gemeinsamen Verteilung von physischen Merkmalen innerhalb und zwischen Gruppen ist die Hautfarbe . Ungefähr 10 % der Varianz der Hautfarbe treten innerhalb von Gruppen auf und ~90 % treten zwischen Gruppen auf (Relethford 2002). Diese Verteilung der Hautfarbe und ihre geografische Musterung – wobei Menschen, deren Vorfahren überwiegend in der Nähe des Äquators lebten, eine dunklere Haut haben als diejenigen mit Vorfahren, die überwiegend in höheren Breiten lebten – weisen darauf hin, dass dieses Attribut unter starkem Selektionsdruck stand . Dunklere Haut scheint in äquatorialen Regionen stark gewählt zu werden, um Sonnenbrand, Hautkrebs, die Photolyse von Folsäure und Schäden an Schweißdrüsen zu verhindern.
Zu verstehen, wie sich die genetische Vielfalt in der menschlichen Bevölkerung auf verschiedene Ebenen der Genexpression auswirkt, ist ein aktives Forschungsgebiet. Während sich frühere Studien auf die Beziehung zwischen DNA-Variation und RNA-Expression konzentrierten, charakterisieren neuere Bemühungen die genetische Kontrolle verschiedener Aspekte der Genexpression, einschließlich Chromatinzustände, Translation und Proteinspiegel. Eine 2007 veröffentlichte Studie ergab, dass 25 % der Gene unterschiedliche Genexpressionsniveaus zwischen Populationen europäischer und asiatischer Abstammung aufwiesen. Als Hauptursache für diesen Unterschied in der Genexpression wurde angenommen, dass SNPs in Genregulationsregionen der DNA sind. Eine andere 2007 veröffentlichte Studie ergab, dass etwa 83% der Gene bei Individuen unterschiedlich stark exprimiert wurden und etwa 17% bei Populationen europäischer und afrikanischer Abstammung.
Wrights Fixationsindex als Maß für die Variation
Der Populationsgenetiker Sewall Wright entwickelte den Fixationsindex (oft mit F ST abgekürzt ), um genetische Unterschiede zwischen Populationen zu messen. Diese Statistik wird häufig in der Taxonomie verwendet, um Unterschiede zwischen zwei beliebigen Populationen zu vergleichen, indem die genetischen Unterschiede zwischen und zwischen Populationen für einzelne Gene oder für viele Gene gleichzeitig gemessen werden. Es wird oft behauptet, dass der Fixationsindex für den Menschen etwa 0,15 beträgt. Dies bedeutet, dass schätzungsweise 85 % der in der gesamten menschlichen Population gemessenen Variation innerhalb derselben Population gefunden werden, und etwa 15 % der Variation treten zwischen Populationen auf. Diese Schätzungen implizieren, dass zwei beliebige Individuen aus unterschiedlichen Populationen einander fast genauso ähnlich sind wie einem Mitglied ihrer eigenen Gruppe. "Die gemeinsame Evolutionsgeschichte der lebenden Menschen hat zu einer hohen Verwandtschaft aller lebenden Menschen geführt, wie zum Beispiel der sehr niedrige Fixationsindex (F ST ) bei lebenden menschlichen Populationen zeigt." Richard Lewontin , der diese Verhältnisse bestätigte, kam daher zu dem Schluss, dass weder "Rasse" noch "Unterart" geeignete oder nützliche Methoden zur Beschreibung menschlicher Populationen seien.
Wright selbst glaubte, dass Werte >0,25 eine sehr große genetische Variation darstellen und dass ein F ST von 0,15–0,25 eine große Variation darstellt. Jedoch treten etwa 5 % der menschlichen Variation zwischen Populationen innerhalb der Kontinente auf, daher wurden in einigen Studien F ST- Werte zwischen kontinentalen Gruppen von Menschen (oder Rassen) von nur 0,1 (oder möglicherweise niedriger) gefunden, was auf ein moderateres genetisches Niveau schließen lässt Variation. Graves (1996) hat dem entgegengehalten, dass F ST nicht als Marker für den Unterartstatus verwendet werden sollte, da die Statistik verwendet wird, um den Differenzierungsgrad zwischen Populationen zu messen, siehe jedoch auch Wright (1978).
Jeffrey Long und Rick Kittles geben in ihrem 2003 erschienenen Aufsatz "Human Genetic Diversity and the Nonexistence of Biological Races" eine lange Kritik an der Anwendung von F ST auf menschliche Populationen. Sie finden, dass die Zahl von 85 % irreführend ist, weil sie impliziert, dass alle menschlichen Populationen im Durchschnitt 85 % der gesamten genetischen Vielfalt enthalten. Sie argumentieren, dass das zugrunde liegende statistische Modell fälschlicherweise gleiche und unabhängige Variationsgeschichten für jede große menschliche Population annimmt. Ein realistischerer Ansatz besteht darin, zu verstehen, dass einige menschliche Gruppen Eltern anderer Gruppen sind und dass diese Gruppen paraphyletische Gruppen für ihre Abstammungsgruppen darstellen. Zum Beispiel ist die menschliche Bevölkerung in Afrika nach der neueren afrikanischen Ursprungstheorie paraphyletisch für alle anderen menschlichen Gruppen, weil sie die Vorfahrengruppe darstellt, von der alle nichtafrikanischen Bevölkerungen abstammen, aber darüber hinaus stammen nichtafrikanische Gruppen nur von einer kleinen nicht repräsentative Stichprobe dieser afrikanischen Bevölkerung. Dies bedeutet, dass alle nichtafrikanischen Gruppen untereinander und mit einigen afrikanischen Gruppen (wahrscheinlich Ostafrikaner) enger verwandt sind als mit anderen, und außerdem stellte die Abwanderung aus Afrika einen genetischen Engpass dar , mit einem Großteil der Vielfalt, die in Afrika existierte und nicht von den auswandernden Gruppen aus Afrika verschleppt wurde. In diesem Szenario weisen menschliche Populationen nicht die gleiche lokale Variabilität auf, sondern eine geringere Diversität, je weiter von Afrika entfernt eine Bevölkerung lebt. Long und Kittles stellen fest, dass statt 85 % der menschlichen genetischen Vielfalt in allen menschlichen Populationen etwa 100 % der menschlichen Vielfalt in einer einzigen afrikanischen Bevölkerung vorhanden sind, während nur etwa 70 % der menschlichen genetischen Vielfalt in einer aus Neuguinea stammenden Bevölkerung existiert. Long und Kittles argumentierten, dass dies immer noch eine globale menschliche Population hervorbringt, die im Vergleich zu anderen Säugetierpopulationen genetisch homogen ist.
Archaische Beimischung
Es gibt eine Hypothese, dass sich anatomisch moderne Menschen mit Neandertalern während des mittleren Paläolithikums gekreuzt haben . Im Mai 2010 legte das Neandertaler-Genomprojekt genetische Beweise dafür vor, dass es wahrscheinlich zu Kreuzungen kam und dass ein kleiner, aber signifikanter Teil, etwa 2–4%, der Neandertaler-Beimischung in der DNA moderner Eurasier und Ozeanier vorhanden ist und in sub -Bevölkerung der Sahara-Afrika.
Es wird angenommen, dass zwischen 4% und 6% des Genoms der Melanesier (vertreten durch die Papua-Neuguineer und Bougainville-Insulaner) von Denisova-Homininen stammen – einer bisher unbekannten Art, die einen gemeinsamen Ursprung mit Neandertalern hat. Es wurde möglicherweise während der frühen Einwanderung der Vorfahren der Melanesier nach Südostasien eingeführt. Diese Interaktionsgeschichte legt nahe, dass Denisova-Menschen einst weit über Ostasien verbreitet waren.
Somit sind Melanesier die am stärksten archaisch vermischte Bevölkerung mit einer Denisovan/Neandertaler-bezogenen Beimischung von ~8%.
In einer 2013 veröffentlichten Studie untersuchte Jeffrey Wall von der University of California ganze Sequenzgenomdaten und fand bei Asiaten im Vergleich zu Europäern höhere Introgressionsraten. Hammeret al. testete die Hypothese, dass zeitgenössische afrikanische Genome Signaturen des Genflusses mit archaischen menschlichen Vorfahren aufweisen, und fanden Beweise für eine archaische Vermischung in den Genomen einiger afrikanischer Gruppen, was darauf hindeutet, dass bescheidene Mengen an Genfluss in Zeit und Raum während der Evolution des anatomisch modernen Menschen weit verbreitet waren .
Kategorisierung der Weltbevölkerung
Neue Daten zur genetischen Variation des Menschen haben die Debatte über eine mögliche biologische Grundlage für die Kategorisierung von Menschen in Rassen neu entfacht. Die meiste Kontroverse dreht sich um die Frage, wie die genetischen Daten zu interpretieren sind und ob die darauf basierenden Schlussfolgerungen stichhaltig sind. Einige Forscher argumentieren, dass die selbstidentifizierte Rasse als Indikator für die geografische Herkunft für bestimmte Gesundheitsrisiken und Medikamente verwendet werden kann .
Obwohl die genetischen Unterschiede zwischen menschlichen Gruppen relativ gering sind, können diese Unterschiede in bestimmten Genen wie Duffy , ABCC11 , SLC24A5 , sogenannte ancestry-informative Marker (AIMs), dennoch verwendet werden, um viele Individuen zuverlässig innerhalb breiter, geografisch basierter Gruppierungen zu lokalisieren. Computeranalysen von Hunderten von polymorphen Loci, die in global verteilten Populationen beprobt wurden, haben beispielsweise die Existenz von genetischen Clustern gezeigt, die grob mit Gruppen in Verbindung gebracht werden, die historisch große kontinentale und subkontinentale Regionen besetzt haben (Rosenberg et al. 2002; Bamshad et al. 2003). .
Einige Kommentatoren haben argumentiert, dass diese Variationsmuster eine biologische Rechtfertigung für die Verwendung traditioneller Rassenkategorien darstellen. Sie argumentieren, dass die kontinentalen Clusterings in etwa der Aufteilung der Menschen in Subsahara-Afrikaner entsprechen ; Europäer , Westasiaten , Zentralasiaten , Südasiaten und Nordafrikaner ; Ostasiaten , Südostasiaten , Polynesier und Indianer ; und andere Bewohner Ozeaniens (Melanesier, Mikronesier & australische Aborigines) (Risch et al. 2002). Andere Beobachter sind anderer Meinung und sagen, dass dieselben Daten traditionelle Vorstellungen von Rassengruppen untergraben (King und Motulsky 2002; Calafell 2003; Tishkoff und Kidd 2004). Sie weisen beispielsweise darauf hin, dass Hauptpopulationen, die als Rassen oder Untergruppen innerhalb von Rassen betrachtet werden, nicht unbedingt ihre eigenen Cluster bilden.
Rassenkategorien werden auch durch die Erkenntnisse untergraben, dass genetische Varianten, die auf eine Region beschränkt sind, innerhalb dieser Region eher selten sind, Varianten, die innerhalb einer Region häufig vorkommen, tendenziell auf der ganzen Welt geteilt werden und die meisten Unterschiede zwischen Individuen, unabhängig davon, ob sie aus der Region stammen, untergraben werden gleiche Region oder unterschiedliche Regionen, sind auf globale Varianten zurückzuführen. Es wurden keine genetischen Varianten gefunden, die innerhalb eines Kontinents oder einer größeren Region fixiert sind und nirgendwo anders gefunden werden.
Da außerdem die genetische Variation des Menschen klinal ist, schließen sich viele Individuen zwei oder mehr kontinentalen Gruppen an. Somit ist die einer Person zugeordnete genetisch bedingte „biogeografische Abstammung“ im Allgemeinen breit gestreut und mit erheblichen Unsicherheiten verbunden (Pfaff et al. 2004).
In vielen Teilen der Welt haben sich Gruppen so vermischt, dass viele Individuen relativ neue Vorfahren aus weit voneinander entfernten Regionen haben. Obwohl genetische Analysen einer großen Anzahl von Loci zu Schätzungen des Prozentsatzes der Vorfahren einer Person aus verschiedenen kontinentalen Populationen führen können (Shriver et al. 2003; Bamshad et al. 2004), können diese Schätzungen eine falsche Unterscheidungskraft der elterlichen Populationen annehmen, da menschliche Gruppen haben im Laufe der Geschichte Partner von lokaler bis kontinentaler Ebene ausgetauscht (Cavalli-Sforza et al. 1994; Hoerder 2002). Selbst bei einer großen Anzahl von Markern sind die Informationen zur Abschätzung der Beimischungsanteile von Individuen oder Gruppen begrenzt, und Schätzungen haben typischerweise breite Konfidenzintervalle (Pfaff et al. 2004).
Genetisches Clustering
Genetische Daten können verwendet werden, um die Bevölkerungsstruktur abzuleiten und Individuen Gruppen zuzuordnen, die oft ihrer selbst identifizierten geographischen Abstammung entsprechen. Jorde und Wooding (2004) argumentierten, dass „die Analyse vieler Loci jetzt ziemlich genaue Schätzungen der genetischen Ähnlichkeit zwischen Individuen und nicht zwischen Populationen liefert. Die Identifizierung nach geografischer Herkunft kann jedoch schnell versagen, wenn man die historische Abstammung betrachtet, die zwischen Einzelpersonen in der Vergangenheit geteilt wurde.
Eine Analyse der autosomalen SNP- Daten aus den Proben des Internationalen HapMap-Projekts (Phase II) und des CEPH Human Genome Diversity Panels wurde 2009 veröffentlicht. Die Studie von 53 Populationen aus den HapMap- und CEPH-Daten (1138 nicht verwandte Individuen) deutete darauf hin, dass die natürliche Auslese beeinflussen kann das menschliche Genom viel langsamer als bisher angenommen, wobei Faktoren wie Migration innerhalb und zwischen Kontinenten die Verbreitung genetischer Variationen stärker beeinflussen. Eine ähnliche Studie, die 2010 veröffentlicht wurde, fand starke genomweite Beweise für die Selektion aufgrund von Veränderungen der Ökoregion, der Ernährung und des Lebensunterhalts, insbesondere in Verbindung mit polaren Ökoregionen, bei der Nahrungssuche und bei einer Ernährung, die reich an Wurzeln und Knollen ist. In einer Studie aus dem Jahr 2016 war die Hauptkomponentenanalyse genomweiter Daten in der Lage, zuvor bekannte Ziele für eine positive Selektion (ohne vorherige Definition von Populationen) sowie eine Reihe neuer Kandidatengene zu gewinnen.
Forensische Anthropologie
Forensische Anthropologen können die Abstammung von Skelettresten beurteilen, indem sie die Skelettmorphologie analysieren und, wenn möglich, genetische und chemische Marker verwenden. Obwohl diese Einschätzungen nie sicher sind, wird die Genauigkeit von Skelettmorphologieanalysen bei der Bestimmung der wahren Abstammung auf 90% geschätzt.

Genfluss und Beimischung
Der Genfluss zwischen zwei Populationen verringert die durchschnittliche genetische Distanz zwischen den Populationen, nur vollständig isolierte menschliche Populationen erfahren keinen Genfluss und die meisten Populationen haben einen kontinuierlichen Genfluss mit anderen benachbarten Populationen, was die für die genetische Variation von Motten beobachtete klinische Verteilung erzeugt. Findet ein Genfluss zwischen gut differenzierten genetischen Populationen statt, wird das Ergebnis als "genetische Beimischung" bezeichnet.
Beimischungskartierung ist eine Technik, die verwendet wird, um zu untersuchen, wie genetische Varianten Unterschiede in den Krankheitsraten zwischen Populationen verursachen. Neuere Beimischungspopulationen, die ihre Vorfahren auf mehrere Kontinente zurückführen, sind gut geeignet, um Gene für Merkmale und Krankheiten zu identifizieren, die sich in der Prävalenz zwischen den Elternpopulationen unterscheiden. Afroamerikanische Populationen standen im Mittelpunkt zahlreicher populationsgenetischer und Beimischungskartierungsstudien, einschließlich Studien zu komplexen genetischen Merkmalen wie der Anzahl der weißen Blutkörperchen, dem Body-Mass-Index, Prostatakrebs und Nierenerkrankungen.
Eine Analyse der phänotypischen und genetischen Variation einschließlich der Hautfarbe und des sozioökonomischen Status wurde in der Bevölkerung der Kapverden durchgeführt, die eine gut dokumentierte Geschichte des Kontakts zwischen Europäern und Afrikanern aufweist. Die Studien zeigten, dass das Beimischungsmuster in dieser Population geschlechtsspezifisch war und dass es eine signifikante Wechselwirkungen zwischen dem sozioökonomischen Status und der Hautfarbe unabhängig von Hautfarbe und Abstammung gibt. Eine andere Studie zeigt ein erhöhtes Risiko für Komplikationen der Graft-versus-Host-Krankheit nach der Transplantation aufgrund genetischer Varianten im humanen Leukozytenantigen (HLA) und Nicht-HLA-Proteinen.
Gesundheit
Unterschiede in der Allelhäufigkeit tragen zu Gruppenunterschieden in der Inzidenz einiger monogener Erkrankungen bei , und sie können zu Unterschieden in der Inzidenz einiger häufiger Erkrankungen beitragen. Für die monogene Krankheiten korreliert die Häufigkeit der ursächliche Allele in der Regel am besten mit Abstammung, ob familiäre (zB Ellis-van Creveld Syndrom unter der Pennsylvania Amish ), ethnischen ( Tay-Sachs - Krankheit unter aschkenasischen jüdischen Bevölkerung) oder geografische (Hämoglobinopathien unter Menschen mit Vorfahren, die in Malariagebieten lebten). In dem Maße, in dem die Abstammung mit rassischen oder ethnischen Gruppen oder Untergruppen übereinstimmt, kann die Inzidenz monogener Krankheiten zwischen den nach Rasse oder ethnischer Zugehörigkeit kategorisierten Gruppen unterschiedlich sein, und Angehörige der Gesundheitsberufe berücksichtigen diese Muster in der Regel bei der Diagnosestellung.
Selbst bei Volkskrankheiten mit zahlreichen genetischen Varianten und Umweltfaktoren weisen die Forscher auf Hinweise auf die Beteiligung unterschiedlich verteilter Allele mit geringen bis mäßigen Auswirkungen hin. Häufig zitierte Beispiele sind Bluthochdruck (Douglas et al. 1996), Diabetes (Gower et al. 2003), Adipositas (Fernandez et al. 2003) und Prostatakrebs (Platz et al. 2000). In keinem dieser Fälle konnte jedoch nachgewiesen werden, dass allelische Variationen in einem Suszeptibilitätsgen einen signifikanten Anteil des Unterschieds in der Krankheitsprävalenz zwischen den Gruppen ausmachen, und die Rolle genetischer Faktoren bei der Generierung dieser Unterschiede bleibt ungewiss (Mountain und Risch 2004).
Einige andere Variationen hingegen sind für den Menschen von Vorteil, da sie bestimmten Krankheiten vorbeugen und die Chance erhöhen, sich an die Umwelt anzupassen. Zum Beispiel Mutation im CCR5- Gen, das vor AIDS schützt . Das CCR5-Gen fehlt aufgrund von Mutationen auf der Zelloberfläche. Ohne das CCR5-Gen an der Oberfläche gibt es für HIV- Viren nichts, woran sie sich festhalten und daran binden können. Daher verringert die Mutation auf dem CCR5-Gen die Wahrscheinlichkeit eines AIDS-Risikos einer Person. Die Mutation in CCR5 ist in bestimmten Gebieten auch recht häufig, wobei mehr als 14% der Bevölkerung die Mutation in Europa und etwa 6–10% in Asien und Nordafrika tragen .
Abgesehen von Mutationen plagen viele Gene, die dem Menschen in der Antike geholfen haben könnten, heute den Menschen. Es wird beispielsweise vermutet, dass Gene, die es dem Menschen ermöglichen, Lebensmittel effizienter zu verarbeiten, diejenigen sind, die heute Menschen anfällig für Fettleibigkeit und Diabetes machen.
Neil Risch von der Stanford University hat vorgeschlagen, dass eine selbstidentifizierte Rasse/ethnische Gruppe ein gültiges Mittel zur Kategorisierung in den USA aus Gründen der öffentlichen Gesundheit und der Politik sein könnte. Eine Veröffentlichung von Noah Rosenbergs Gruppe aus dem Jahr 2002 macht eine ähnliche Behauptung: „Die Struktur der menschlichen Populationen ist in verschiedenen epidemiologischen Kontexten relevant Nebenwirkungen von Arzneimitteln variieren je nach Bevölkerungsgruppe. Darüber hinaus können Informationen über die Herkunftspopulation eines Patienten dem Gesundheitspersonal Informationen über das Risiko liefern, wenn die direkten Krankheitsursachen unbekannt sind.“ Doch im Jahr 2018 Noah Rosenberg veröffentlichte eine Studie argumentiert gegen genetisch essentialistischen Vorstellungen von Gesundheit Unterschieden zwischen den Populationen unter Angabe Umweltvarianten eine wahrscheinlichere Ursache sind Interpretieren polygener Partituren, polygener Anpassung und menschliche phänotypische Unterschiede
Genomprojekte
Humangenomprojekte sind wissenschaftliche Vorhaben , die die Struktur des menschlichen Genoms bestimmen oder untersuchen . Das Human Genome Project war ein wegweisendes Genomprojekt.
Siehe auch
- Rasse und Genetik
- Archäogenetik
- Evolutionäre Genetik des Menschen
- Multiregionale Hypothese
- Aktuelle Single-Origin-Hypothese
- Isolation durch Distanz
- Genealogischer DNA-Test
- Y-Chromosom-Haplogruppen in Populationen der Welt
- Neurodiversität
Regional:
- Genetische Geschichte Europas
- Genetische Geschichte Südasiens
- Afrikanische Beimischung in Europa
- Genetische Geschichte der indigenen Völker Amerikas
- Genetische Geschichte der britischen Inseln
Projekte:
Verweise
Weiterlesen
- Rasse, Ethnizität (Oktober 2005). "Die Verwendung von Rassen-, ethnischen und Ahnenkategorien in der Humangenetikforschung" . Amerikanisches Journal für Humangenetik . 77 (4): 519–32. doi : 10.1086/491747 . PMC 1275602 . PMID 16175499 .
- Altmüller J, Palmer LJ, Fischer G, Scherb H, Wjst M (November 2001). „Genomweite Scans komplexer menschlicher Krankheiten: Eine wahre Verbindung ist schwer zu finden“ . Amerikanisches Journal für Humangenetik . 69 (5): 936–50. doi : 10.1086/324069 . PMC 1274370 . PMID 11565063 .
- Aoki K (2002). „Sexuelle Selektion als Ursache für die Variation der menschlichen Hautfarbe: Darwins Hypothese revisited“. Annalen der Humanbiologie . 29 (6): 589–608. doi : 10.1080/030144602100019144 . PMID 12573076 . S2CID 22703861 .
- Bamshad M, Wooding S, Salisbury BA, Stephens JC (August 2004). „Dekonstruktion der Beziehung zwischen Genetik und Rasse“. Natur Bewertungen. Genetik . 5 (8): 598–609. doi : 10.1038/nrg1401 . PMID 15266342 . S2CID 12378279 . Nachdruck-zip
- Bamshad M, Wooding SP (Februar 2003). „Signaturen der natürlichen Selektion im menschlichen Genom“. Natur Bewertungen. Genetik . 4 (2): 99–111. doi : 10.1038/nrg999 . PMID 12560807 . S2CID 13722452 .
- Cann RL, Stoneking M, Wilson AC (1987). „Mitochondriale DNA und die menschliche Evolution“. Natur . 325 (6099): 31–36. Bibcode : 1987Natur.325...31C . doi : 10.1038/325031a0 . PMID 3025745 . S2CID 4285418 .
- Cardon LR, Abecasis GR (März 2003). "Verwendung von Haplotyp-Blöcken zur Kartierung menschlicher Loci komplexer Merkmale" (PDF) . Trends in der Genetik . 19 (3): 135–40. doi : 10.1016/S0168-9525(03)00022-2 . PMID 12615007 .
- Cavalli-Sforza LL, Feldman MW (März 2003). „Die Anwendung molekulargenetischer Ansätze auf das Studium der menschlichen Evolution“. Naturgenetik . 33 Zusatz (3s): 266–75. doi : 10.1038/ng1113 . PMID 12610536 . S2CID 8314161 .
- Collins FS (November 2004). „Was wir zu Beginn der Genom-Ära über ‚Rasse‘, ‚Ethnizität‘, Genetik und Gesundheit wissen und was wir nicht wissen“ . Naturgenetik . 36 (11 Ergänzung): S13–15. doi : 10.1038/ng1436 . PMID 15507997 . S2CID 26968169 .
- Collins FS, Green ED, Guttmacher AE, Guyer MS (April 2003). „Eine Vision für die Zukunft der Genomforschung“. Natur . 422 (6934): 835–47. Bibcode : 2003Natur.422..835C . doi : 10.1038/nature01626 . PMID 12695777 . S2CID 205209730 .
- Ebersberger I, Metzler D, Schwarz C, Pääbo S (Juni 2002). „Genomweiter Vergleich von DNA-Sequenzen zwischen Mensch und Schimpanse“ . Amerikanisches Journal für Humangenetik . 70 (6): 1490–97. doi : 10.1086/340787 . PMC 379137 . PMID 11992255 .
- Edwards AW (August 2003). „Menschliche genetische Vielfalt: Lewontins Irrtum“. BioEssays . 25 (8): 798–801. doi : 10.1002/bies.10315 . PMID 12879450 .
- Foster MW, Sharp RR (Oktober 2004). „Beyond Race: Hin zu einer Gesamtgenom-Perspektive auf menschliche Populationen und genetische Variation“. Natur Bewertungen. Genetik . 5 (10): 790–96. doi : 10.1038/nrg1452 . PMID 15510170 . S2CID 25764082 .
- Foster MW, Sharp RR, Freeman WL, Chino M, Bernsten D, Carter TH (Juni 1999). "Die Rolle der Gemeinschaftsbewertung bei der Bewertung der Risiken der Erforschung der genetischen Variation beim Menschen" . Amerikanisches Journal für Humangenetik . 64 (6): 1719–27. doi : 10.1086/302415 . PMC 1377916 . PMID 10330360 .
- Gabriel SB, Schaffner SF, Nguyen H, Moore JM, Roy J, Blumenstiel B, Higgins J, DeFelice M, Lochner A, Faggart M, Liu-Cordero SN, Rotimi C, Adeyemo A, Cooper R, Ward R, Lander ES, Daly MJ, Altschuler D (Juni 2002). „Die Struktur von Haplotyp-Blöcken im menschlichen Genom“. Wissenschaft . 296 (5576): 2225–29. Bibcode : 2002Sc...296.2225G . doi : 10.1126/science.1069424 . PMID 12029063 . S2CID 10069634 .
- Harding RM, Healy E, Ray AJ, Ellis NS, Flanagan N, Todd C, Dixon C, Sajantila A, Jackson IJ, Birch-Machin MA, Rees JL (April 2000). „Nachweis für variable selektive Drücke bei MC1R“ . Amerikanisches Journal für Humangenetik . 66 (4): 1351–61. doi : 10.1086/302863 . PMC 1288200 . PMID 10733465 .
- Ingman M, Kaessmann H, Pääbo S, Gyllensten U (Dezember 2000). „Mitochondriale Genomvariation und der Ursprung des modernen Menschen“. Natur . 408 (6813): 708–13. Bibcode : 2000Natur.408..708I . doi : 10.1038/35047064 . PMID 11130070 . S2CID 52850476 .
- Das Internationale Hapmap-Konsortium (Dezember 2003). "Das internationale HapMap-Projekt". Natur . 426 (6968): 789–96. Bibcode : 2003Natur.426..789G . doi : 10.1038/nature02168 . hdl : 2027.42/62838 . PMID 14685227 . S2CID 4387110 .
- Das Internationale Hapmap-Konsortium (Juni 2004). "Integration von Ethik und Wissenschaft in das Internationale HapMap-Projekt" . Natur Bewertungen. Genetik . 5 (6): 467–75. doi : 10.1038/nrg1351 . PMC 2271136 . PMID 15153999 .
- Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, et al. (Februar 2001). "Erste Sequenzierung und Analyse des menschlichen Genoms" . Natur . 409 (6822): 860–921. Bibcode : 2001Natur.409..860L . doi : 10.1038/35057062 . PMID 11237011 .
- Jorde LB, Bamshad M, Rogers AR (Februar 1998). "Mitochondriale und nukleare DNA-Marker verwenden, um die menschliche Evolution zu rekonstruieren" (PDF) . BioEssays . 20 (2): 126–36. doi : 10.1002/(SICI)1521-1878(199802)20:2<126::AID-BIES5>3.0.CO;2-R . PMID 9631658 . Archiviert vom Original (PDF) am 28. November 2007 . Abgerufen am 28. Oktober 2007 .
- Jorde LB, Watkins WS, Bamshad MJ, Dixon ME, Ricker CE, Seielstad MT, Batzer MA (März 2000). „Die Verteilung der menschlichen genetischen Vielfalt: ein Vergleich von mitochondrialen, autosomalen und Y-Chromosom-Daten“ . Amerikanisches Journal für Humangenetik . 66 (3): 979–88. doi : 10.1086/302825 . PMC 1288178 . PMID 10712212 .
- Jorde LB, Watkins WS, Kere J, Nyman D, Eriksson AW (2000). "Genkartierung in isolierten Populationen: neue Rollen für alte Freunde?". Menschliche Vererbung . 50 (1): 57–65. doi : 10.1159/000022891 . PMID 10545758 . S2CID 26960216 .
- Kaessmann H, Heissig F, von Haeseler A, Pääbo S (Mai 1999). „DNA-Sequenzvariation in einer nicht-kodierenden Region geringer Rekombination auf dem menschlichen X-Chromosom“. Naturgenetik . 22 (1): 78–81. doi : 10.1038/8785 . PMID 10319866 . S2CID 9153915 .
- Kaessmann H, Wiebe V, Weiss G, Pääbo S (Februar 2001). "DNA-Sequenzen von Menschenaffen zeigen eine reduzierte Vielfalt und eine Ausdehnung beim Menschen". Naturgenetik . 27 (2): 155–56. doi : 10.1038/84773 . PMID 11175781 . S2CID 19384784 .
- Keita SO, Kittles RA (1997). „Die Beständigkeit des rassischen Denkens und der Mythos der Rassendivergenz“. US-amerikanischer Anthropologe . 99 (3): 534–44. doi : 10.1525/aa.1997.99.3.534 .
- Marks J (1995). Biodiversität des Menschen: Gene, Rasse und Geschichte . Aldine-Transaktion . ISBN 978-0-202-02033-4.
- Berg JL, Risch N (November 2004). „Bewertung genetischer Beiträge zu phänotypischen Unterschieden zwischen ‚rassischen‘ und ‚ethnischen‘ Gruppen“ . Naturgenetik . 36 (11 Ergänzung): S48–53. doi : 10.1038/ng1456 . PMID 15508003 .
- Pääbo S (Januar 2003). "Das Mosaik, das unser Genom ist" . Natur . 421 (6921): 409–12. Bibcode : 2003Natur.421..409P . doi : 10.1038/nature01400 . PMID 12540910 .
- Ramachandran S, Deshpande O, Roseman CC, Rosenberg NA, Feldman MW, Cavalli-Sforza LL (November 2005). "Unterstützung aus der Beziehung von genetischer und geografischer Distanz in menschlichen Populationen für einen seriellen Gründereffekt mit Ursprung in Afrika" . Proceedings of the National Academy of Sciences of the United States of America . 102 (44): 15942–47. Bibcode : 2005PNAS..10215942R . doi : 10.1073/pnas.05076111102 . PMC 1276087 . PMID 16243969 .
- Relethford JH (August 2002). "Aufteilung der globalen genetischen Vielfalt des Menschen basierend auf Kraniometrie und Hautfarbe". Amerikanische Zeitschrift für physikalische Anthropologie . 118 (4): 393–98. CiteSeerX 10.1.1.473.5972 . doi : 10.1002/ajpa.10079 . PMID 12124919 .
- Sankar P, Cho MK (November 2002). "Genetik. Auf dem Weg zu einem neuen Vokabular der menschlichen genetischen Variation" . Wissenschaft . 298 (5597): 1337–38. doi : 10.1126/science.1074447 . PMC 2271140 . PMID 12434037 .
- Sankar P, Cho MK, Condit CM, Hunt LM, Koenig B, Marshall P, Lee SS, Spicer P (Juni 2004). "Genforschung und gesundheitliche Disparitäten" . JAMA . 291 (24): 2985–89. doi : 10.1001/jama.291.24.2985 . PMC 2271142 . PMID 15213210 .
- Serre D, Pääbo S (September 2004). „Beweise für Gradienten der menschlichen genetischen Vielfalt innerhalb und zwischen Kontinenten“ . Genomforschung . 14 (9): 1679–85. doi : 10.1011/gr.2529604 . PMC 515312 . PMID 15342553 .
- Templeton AR (1998). „Menschliche Rassen: Eine genetische und evolutionäre Perspektive“. US-amerikanischer Anthropologe . 100 (3): 632–50. doi : 10.1525/aa.1998.100.3.632 .
- Weiss KM (1998). „Mit der menschlichen Variation klarkommen“. Jährliche Überprüfung der Anthropologie . 27 : 273–300. doi : 10.1146/annurev.anthro.27.1.273 .
- Weiss KM, Terwilliger JD (Oktober 2000). "Wie viele Krankheiten braucht es, um ein Gen mit SNPs zu kartieren?". Naturgenetik . 26 (2): 151–57. doi : 10.1038/79866 . PMID 11017069 . S2CID 685795 .
- Yu N, Jensen-Seaman MI, Chemnick L, Kidd JR, Deinard AS, Ryder O, Kidd KK, Li WH (August 2003). „Niedrige Nukleotid-Diversität bei Schimpansen und Bonobos“ . Genetik . 164 (4): 1511-18. doi : 10.1093/Genetik/164.4.1511 . PMC 1462640 . PMID 12930756 .
- Zietkiewicz E, Yotova V, Gehl D, Wambach T, Arrieta I, Batzer M, Cole DE, Hechtman P, Kaplan F, Modiano D, Moisan JP, Michalski R, Labuda D (November 2003). "Haplotypen im Dystrophin-DNA-Segment weisen auf einen mosaikartigen Ursprung der modernen menschlichen Vielfalt hin" . Amerikanisches Journal für Humangenetik . 73 (5): 994–1015. doi : 10.1086/378777 . PMC 1180505 . PMID 14513410 .
- Pennisi E (Dezember 2007). "Durchbruch des Jahres. Menschliche genetische Variation" . Wissenschaft . 318 (5858): 1842–43. doi : 10.1126/science.318.5858.1842 . PMID 18096770 .
- Ramachandran S, Tang H, Gutenkunst RN, Bustamante CD (2010). „Genetik und Genomik der menschlichen Bevölkerungsstruktur“. In Speicher MR, Antonarakis SE, Motulsky AG (Hrsg.). Vogel und Motulskys Humangenetik: Probleme und Ansätze (4. Aufl.). Springer . ISBN 978-3-540-37653-8.