Human-Leukozyten-Antigen - Human leukocyte antigen
| Human-Leukozyten-Antigen | |
|---|---|
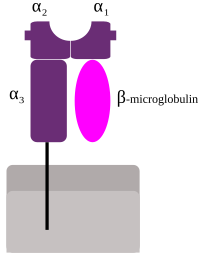 Schematische Darstellung der MHC-Klasse I
| |
| Bezeichner | |
| Symbol | HLA |
| InterPro | IPR037055 |
| Membranom | 63 |
Das humane Leukozytenantigen ( HLA )-System oder -Komplex ist ein Komplex von Genen auf Chromosom 6 beim Menschen, die für die Regulierung des Immunsystems verantwortliche Zelloberflächenproteine kodieren . Das HLA-System ist auch als die menschliche Version des Haupthistokompatibilitätskomplexes (MHC) bekannt, der bei vielen Tieren vorkommt.
Mutationen in HLA-Genen können mit Autoimmunerkrankungen wie Typ-I-Diabetes und Zöliakie in Verbindung gebracht werden . Der HLA-Genkomplex befindet sich auf einer 3 Mbp- Strecke innerhalb von Chromosom 6, p-Arm bei 21,3. HLA-Gene sind hochgradig polymorph , das heißt, sie haben viele verschiedene Allele , die es ihnen ermöglichen, das adaptive Immunsystem zu verfeinern . Die von bestimmten Genen kodierten Proteine werden aufgrund ihrer historischen Entdeckung als Faktoren bei Organtransplantationen auch als Antigene bezeichnet .
HLAs, die der MHC-Klasse I ( A , B und C ) entsprechen, die alle die HLA-Klasse-1-Gruppe sind, präsentieren Peptide aus dem Inneren der Zelle. Ist die Zelle beispielsweise mit einem Virus infiziert, bringt das HLA-System Bruchstücke des Virus an die Zelloberfläche, damit die Zelle vom Immunsystem zerstört werden kann. Diese Peptide werden aus verdauten Proteinen hergestellt, die in den Proteasomen abgebaut werden . Im Allgemeinen sind diese speziellen Peptide kleine Polymere mit einer Länge von etwa 8–10 Aminosäuren . Fremde Antigene, die von MHC Klasse I präsentiert werden, ziehen T-Lymphozyten an, die Killer-T-Zellen genannt werden (auch als CD8- positive oder zytotoxische T-Zellen bezeichnet), die Zellen zerstören. Einige neue Arbeiten haben vorgeschlagen, dass Antigene mit einer Länge von mehr als 10 Aminosäuren, 11–14 Aminosäuren, auf MHC I präsentiert werden können, was eine zytotoxische T-Zell-Antwort hervorruft. MHC-Klasse-I-Proteine assoziieren mit β2-Mikroglobulin , das im Gegensatz zu den HLA-Proteinen von einem Gen auf Chromosom 15 kodiert wird .
HLAs, die der MHC-Klasse II ( DP , DM , DO , DQ und DR ) entsprechen, präsentieren Antigene von außerhalb der Zelle an T-Lymphozyten. Diese speziellen Antigene stimulieren die Vermehrung von T-Helferzellen (auch CD4- positive T-Zellen genannt), die wiederum Antikörper- produzierende B-Zellen stimulieren , Antikörper gegen dieses spezifische Antigen zu produzieren. Selbstantigene werden durch regulatorische T-Zellen unterdrückt . Es ist schwierig vorherzusagen, welche (Fragmente von) Antigenen dem Immunsystem von einem bestimmten HLA-Typ präsentiert werden, aber die damit verbundene Technologie verbessert sich.
HLAs, die der MHC-Klasse III entsprechen, codieren Komponenten des Komplementsystems .
HLAs haben andere Rollen. Sie sind wichtig für die Abwehr von Krankheiten. Sie sind die Hauptursache für die Abstoßung von Organtransplantationen . Sie können vor Krebs schützen oder nicht schützen (wenn sie durch eine Infektion herunterreguliert werden). HLA kann auch mit der Wahrnehmung des Geruchs anderer Menschen durch die Menschen zusammenhängen und an der Partnerwahl beteiligt sein, da mindestens eine Studie eine niedriger als erwartete HLA-Ähnlichkeit zwischen Ehepartnern in einer isolierten Gemeinschaft gefunden hat.
Abgesehen von den Genen, die für die sechs wichtigsten Antigen-präsentierenden Proteine kodieren, befinden sich im HLA-Komplex viele andere Gene, von denen viele an der Immunfunktion beteiligt sind. Die Vielfalt der HLAs in der menschlichen Bevölkerung ist ein Aspekt der Krankheitsabwehr, und daher ist die Wahrscheinlichkeit, dass zwei nicht verwandte Individuen mit identischen HLA-Molekülen an allen Loci vorhanden sind, extrem gering. HLA-Gene wurden in der Vergangenheit als Ergebnis der Fähigkeit identifiziert, erfolgreich Organe zwischen HLA-ähnlichen Individuen zu transplantieren.
Funktionen
Die Proteine , kodiert durch HLAs sind jene auf dem äußeren Teil der Körperzellen , die (in der Tat) einzigartig sind für diese Person sind. Das Immunsystem verwendet die HLAs, um Eigenzellen und Nicht-Eigenzellen zu unterscheiden. Jede Zelle, die den HLA-Typ dieser Person anzeigt, gehört zu dieser Person und ist daher kein Eindringling.
Bei Infektionskrankheiten
Wenn ein fremder Krankheitserreger in den Körper eindringt, verschlingen spezifische Zellen, die als Antigen-präsentierende Zellen (APCs) bezeichnet werden, den Krankheitserreger durch einen Prozess, der Phagozytose genannt wird . Proteine des Erregers werden in kleine Stücke ( Peptide ) verdaut und auf HLA-Antigene (genauer gesagt MHC-Klasse II ) geladen . Sie werden dann von den angezeigten Antigen-präsentierenden Zellen zu CD4 + T - Helferzellen , die dann eine Vielzahl von Effekten und Zell - Zell - Wechselwirkungen erzeugen , um den Erreger zu eliminieren.
Durch einen ähnlichen Prozess werden Proteine (sowohl native als auch fremde, wie die Proteine des Virus), die in den meisten Zellen produziert werden, auf HLAs (genauer gesagt MHC-Klasse I ) auf der Zelloberfläche präsentiert. Infizierte Zellen können von CD8+ T-Zellen erkannt und zerstört werden .
Das nebenstehende Bild zeigt ein Stück eines giftigen bakteriellen Proteins (SEI-Peptid), das im Bindespalt des HLA-DR1-Moleküls gebunden ist. In der Abbildung weit unten, einer anderen Ansicht, sieht man ein ganzes DQ mit einem gebundenen Peptid in einer ähnlichen Spalte von der Seite gesehen. In diese "Schlitze" passen krankheitsbezogene Peptide, ähnlich wie eine Hand in einen Handschuh passt. Wenn sie gebunden sind, werden Peptide T-Zellen präsentiert. T-Zellen benötigen eine Präsentation über MHC-Moleküle, um fremde Antigene zu erkennen – eine Voraussetzung, die als MHC-Restriktion bekannt ist . T-Zellen haben Rezeptoren, die B-Zell-Rezeptoren ähnlich sind, und jede T-Zelle erkennt nur wenige MHC-Klasse-II-Peptid-Kombinationen. Sobald eine T-Zelle ein Peptid innerhalb eines MHC-Klasse-II-Moleküls erkennt, kann sie B-Zellen stimulieren, die dasselbe Molekül auch in ihren B-Zell-Rezeptoren erkennen. Somit helfen T-Zellen den B-Zellen, Antikörper gegen dieselben fremden Antigene zu bilden. Jedes HLA kann viele Peptide binden, und jede Person hat 3 HLA-Typen und kann 4 Isoformen von DP, 4 Isoformen von DQ und 4 Isoformen von DR (2 von DRB1 und 2 von DRB3, DRB4 oder DRB5) für insgesamt 12 Isoformen. Bei solchen Heterozygoten ist es für krankheitsbezogene Proteine schwierig, der Detektion zu entgehen.
Bei Transplantatabstoßung
Jede Zelle, die einen anderen HLA-Typ aufweist, ist "nicht-eigen" und wird vom Immunsystem des Körpers als Eindringling angesehen, was zur Abstoßung des Gewebes führt, das diese Zellen trägt. Dies ist besonders bei transplantiertem Gewebe wichtig, da es zu einer Transplantatabstoßung kommen kann . Aufgrund der Bedeutung von HLA bei der Transplantation gehören die HLA-Loci zu den am häufigsten durch Serologie und PCR typisierten. Es hat sich gezeigt, dass eine hochauflösende HLA-Typisierung (HLA-A, HLA-B, HLA-C, HLA-DRB1, HLA-DQB1 und HLA-DPB1) bei der Transplantation relevant sein kann, um eine vollständige Übereinstimmung zu identifizieren, selbst wenn der Spender verbunden.
| HLA-Allel | Krankheiten mit erhöhtem Risiko | Relatives Risiko |
|---|---|---|
| HLA-B27 | Spondylitis ankylosans | 12 |
| Reaktive Arthritis | 14 | |
| Akute anteriore Uveitis | fünfzehn | |
| HLA-B47 | 21-Hydroxylase-Mangel | fünfzehn |
| HLA-DR2 | Systemischer Lupus erythematodes | 2 bis 3 |
| HLA-DR3 | Autoimmunhepatitis | 14 |
| Primäres Sjögren-Syndrom | 10 | |
| Diabetes mellitus Typ 1 | 5 | |
| Systemischer Lupus erythematodes | 2 bis 3 | |
| HLA-DR4 | Rheumatoide Arthritis | 4 |
| Diabetes mellitus Typ 1 | 6 | |
| HLA-DR3 und -DR4 kombiniert |
Diabetes mellitus Typ 1 | fünfzehn |
| HLA-DQ2 und HLA-DQ8 | Zöliakie | 7 |
Bei Autoimmunität
HLA-Typen werden vererbt, und einige von ihnen sind mit Autoimmunerkrankungen und anderen Krankheiten verbunden. Menschen mit bestimmten HLA-Antigenen entwickeln mit größerer Wahrscheinlichkeit bestimmte Autoimmunerkrankungen wie Typ-I-Diabetes , Spondylitis ankylosans , rheumatoide Arthritis , Zöliakie , SLE (systemischer Lupus erythematodes), Myasthenia gravis , Einschlusskörpermyositis , Sjögren-Syndrom und Narkolepsie . Die HLA-Typisierung hat zu einer gewissen Verbesserung und Beschleunigung der Diagnose von Zöliakie und Typ-1-Diabetes geführt; Damit die DQ2-Typisierung jedoch nützlich ist, ist entweder eine hochauflösende B1*-Typisierung (Auflösung von *02:01 von *02:02), DQA1*-Typisierung oder DR- Serotypisierung erforderlich . Die derzeitige Serotypisierung kann DQ8 in einem Schritt auflösen. Die HLA-Typisierung bei Autoimmunität wird zunehmend als Werkzeug in der Diagnose verwendet. Bei Zöliakie ist es das einzige wirksame Mittel, um gefährdete Verwandte ersten Grades von nicht gefährdeten Verwandten zu unterscheiden, bevor manchmal irreversible Symptome wie Allergien und sekundäre Autoimmunerkrankungen auftreten.
Bei Krebs
Einige HLA-vermittelte Krankheiten sind direkt an der Förderung von Krebs beteiligt. Glutensensitive Enteropathie ist mit einer erhöhten Prävalenz von Enteropathie-assoziierten T-Zell-Lymphomen verbunden, und DR3-DQ2-Homozygoten gehören mit fast 80 % der Fälle von glutensensitiven Enteropathie-assoziierten T-Zell-Lymphomen zur Gruppe mit dem höchsten Risiko. Häufiger jedoch spielen HLA-Moleküle eine schützende Rolle, indem sie Zunahmen von Antigenen erkennen, die aufgrund niedriger Spiegel im Normalzustand nicht vertragen werden. Abnormale Zellen könnten für die Apoptose anvisiert werden, von der angenommen wird, dass sie viele Krebsarten vor der Diagnose vermittelt.
In der Partnerwahl
Es gibt Hinweise auf eine nicht-zufällige Partnerwahl in Bezug auf bestimmte genetische Merkmale. Dies hat zu einem Feld geführt, das als genetisches Matchmaking bekannt ist .
Einstufung
MHC-Klasse-I-Proteine bilden auf den meisten kernhaltigen Zellen des Körpers einen funktionellen Rezeptor.
Es gibt 3 Major- und 3 Minor-MHC-Klasse-I-Gene in HLA.
Major MHC Klasse I
Nebengene sind HLA-E , HLA-F und HLA-G . β 2 -Mikroglobulin bindet an Haupt- und Nebengen- Untereinheiten, um ein Heterodimer zu erzeugen
Es gibt 3 Haupt- und 2 Nebenproteine der MHC-Klasse II, die von HLA kodiert werden. Die Gene der Klasse II verbinden sich zu heterodimeren (αβ) Proteinrezeptoren, die typischerweise auf der Oberfläche von Antigen-präsentierenden Zellen exprimiert werden .
Wichtige MHC-Klasse-II-Proteine kommen nur auf Antigen-präsentierenden Zellen , B-Zellen und T-Zellen vor .
-
HLA-DP
- α-Kette, kodiert durch den HLA-DPA1-Locus
- β-Kette, kodiert durch den HLA-DPB1- Locus
- HLA-DQ
-
HLA-DR
- α-Kette, kodiert durch den HLA-DRA-Locus
- 4 β-Ketten (nur 3 pro Person möglich), kodiert von HLA-DRB1, DRB3, DRB4, DRB5 Loci
Die anderen MHC-Klasse-II-Proteine, DM und DO, werden bei der internen Verarbeitung von Antigenen verwendet, indem die von Pathogenen erzeugten antigenen Peptide auf die HLA-Moleküle der Antigen-präsentierenden Zelle geladen werden .
Nomenklatur
Moderne HLA-Allele werden typischerweise mit einer Vielzahl von Detaillierungsebenen notiert. Die meisten Bezeichnungen beginnen mit HLA- und dem Locus-Namen, dann * und einer (gerade) Anzahl von Ziffern, die das Allel spezifizieren. Die ersten beiden Ziffern geben eine Gruppe von Allelen an, auch Supertypen genannt. Ältere Typisierungsmethoden konnten Allele oft nicht vollständig unterscheiden und blieben daher auf dieser Ebene stehen. Die dritte bis vierte Ziffer geben ein nicht-synonymes Allel an. Die Ziffern fünf bis sechs bezeichnen alle synonymen Mutationen innerhalb des Kodierungsrahmens des Gens. Die siebte und achte Ziffer unterscheiden Mutationen außerhalb der kodierenden Region. Buchstaben wie L, N, Q oder S können auf die Bezeichnung eines Allels folgen, um ein Expressionsniveau oder andere nicht genomische Daten, die darüber bekannt sind, anzugeben. Somit kann ein vollständig beschriebenes Allel bis zu 9 Ziffern lang sein, ohne HLA-Präfix und Locus-Notation.
Variabilität
MHC-Loci sind einige der genetisch am variabelsten kodierenden Loci bei Säugetieren, und die humanen HLA-Loci sind keine Ausnahmen. Trotz der Tatsache, dass die menschliche Bevölkerung im Laufe ihrer Geschichte mehrmals eine Verengung durchgemacht hat, die in der Lage war, viele Loci zu reparieren, scheinen die HLA-Loci eine solche Verengung mit vielen Variationen überlebt zu haben. Von den 9 oben erwähnten Loci behielten die meisten ein Dutzend oder mehr Allelgruppen für jeden Locus bei, eine weitaus stärker erhaltene Variation als die überwiegende Mehrheit der menschlichen Loci. Dies stimmt mit einem heterozygoten oder ausgleichenden Selektionskoeffizienten für diese Loci überein . Darüber hinaus gehören einige HLA-Loci zu den sich am schnellsten entwickelnden kodierenden Regionen im menschlichen Genom. Ein Diversifizierungsmechanismus wurde in der Studie von Amazonas-Stämmen in Südamerika festgestellt, die anscheinend eine intensive Genkonversion zwischen variablen Allelen und Loci innerhalb jeder HLA-Genklasse durchlaufen haben . Weniger häufig wurden produktive Rekombinationen über längere Reichweiten durch HLA-Gene festgestellt, die chimäre Gene produzieren.
Sechs Loci haben über 100 Allele, die in der menschlichen Bevölkerung nachgewiesen wurden. Von diesen sind HLA B und HLA DRB1 die variabelsten. Die Anzahl der ermittelten Allele (Stand 2012) ist in der folgenden Tabelle aufgeführt. Um diese Tabelle zu interpretieren, muss berücksichtigt werden, dass ein Allel eine Variante der Nukleotid(DNA)-Sequenz an einem Locus ist, so dass sich jedes Allel von allen anderen Allelen in mindestens einer Position (Single Nucleotide Polymorphism, SNP) unterscheidet. Die meisten dieser Veränderungen führen zu einer Veränderung der Aminosäuresequenzen, die zu leichten bis großen funktionellen Unterschieden im Protein führt.
Es gibt Probleme, die diese Variation einschränken. Bestimmte Allele wie DQA1*05:01 und DQA1*05:05 kodieren Proteine mit identisch verarbeiteten Produkten. Andere Allele wie DQB1*0201 und DQB1*0202 produzieren funktionell ähnliche Proteine. Bei Klasse II (DR, DP und DQ) neigen Aminosäurevarianten innerhalb der Peptidbindungsspalte des Rezeptors dazu, Moleküle mit unterschiedlicher Bindungsfähigkeit zu produzieren.
Die Genhäufigkeiten der häufigsten Allele (>5%) von HLA-A, -B, -C und HLA-DPA1, -DPB1, -DQA1, -DQB1 und -DRB1 aus Südamerika wurden jedoch von der Typisierung und Sequenzierung in Studien zur genetischen Vielfalt sowie Fälle und Kontrollen. Darüber hinaus wurden Informationen zu den Allelfrequenzen der HLA-I- und HLA-II-Gene für die europäische Bevölkerung zusammengestellt. In beiden Fällen zeigt die Verteilung der Allelhäufigkeiten eine regionale Variation im Zusammenhang mit der Geschichte der Populationen.
Tabellen der Variantenallele
Anzahl der Variantenallele an Klasse-I-Loci gemäß IMGT-HLA-Datenbank, Stand Oktober 2018:
| MHC-Klasse I | |
|---|---|
| Ort | # |
| Hauptantigene | |
| HLA A | 4.340 |
| HLA B | 5,212 |
| HLA C | 3.930 |
| Geringe Antigene | |
| HLA E | 27 |
| HLA F | 31 |
| HLA G | 61 |
Anzahl der Varianten-Allele an Klasse-II-Loci (DM, DO, DP, DQ und DR):
| MHC-Klasse II | ||||
|---|---|---|---|---|
| HLA | -A1 | -B1 | -B3 bis -B5 1 | Theor. möglich |
| Ort | # | # | # | Kombinationen |
| DM- | 7 | 13 | 91 | |
| TUN- | 12 | 13 | 156 | |
| DP- | 67 | 1.014 | 16.036 | |
| DQ- | 95 | 1.257 | 34.528 | |
| DR- | 7 | 2.593 | 312 | 11.431 |
| 1 DRB3, DRB4, DRB5 sind beim Menschen unterschiedlich vorhanden | ||||
Variantentyp Sequenzmerkmal (SFVT)
Die große Variabilität der HLA-Gene stellt erhebliche Herausforderungen bei der Untersuchung der Rolle genetischer Variationen von HLA bei Krankheiten dar. Krankheitsassoziationsstudien behandeln typischerweise jedes HLA-Allel als eine einzelne vollständige Einheit, die die mit der Krankheit verbundenen Teile des Moleküls nicht beleuchtet. Karp DR et al. beschreibt einen neuartigen Ansatz des Sequenzmerkmalsvariantentyps (SFVT) für die genetische Analyse von HLA, der HLA-Proteine in biologisch relevante kleinere Sequenzmerkmale (SFs) und deren Variantentypen (VTs) kategorisiert. Sequenzmerkmale sind Kombinationen von Aminosäurestellen, die basierend auf Strukturinformationen (zB Beta-Faltblatt 1), funktionellen Informationen (zB Peptidantigenbindung) und Polymorphismus definiert sind. Diese Sequenzmerkmale können in der linearen Sequenz überlappend und kontinuierlich oder diskontinuierlich sein. Variantentypen für jedes Sequenzmerkmal werden basierend auf allen bekannten Polymorphismen in dem beschriebenen HLA-Locus definiert. Die SFVT-Kategorisierung von HLA wird in der genetischen Assoziationsanalyse angewendet, so dass die Wirkungen und Rollen der Epitope identifiziert werden können, die von mehreren HLA-Allelen geteilt werden. Sequenzmerkmale und deren Variantentypen wurden für alle klassischen HLA-Proteine beschrieben; das internationale Repository von HLA SFVTs wird in der IMGT/HLA-Datenbank geführt. Ein Tool zur Umwandlung von HLA-Allelen in ihre SFVT-Komponenten finden Sie auf der Website des Immunology Database and Analysis Portal (ImmPort).
Häufige, gut dokumentierte und seltene Allele
Obwohl die Zahl der identifizierten einzelnen HLA-Allele groß ist, scheinen ungefähr 40% dieser Allele einzigartig zu sein, da sie nur bei einzelnen Individuen identifiziert wurden. Ungefähr ein Drittel der Allele wurde bei nicht verwandten Personen mehr als dreimal gemeldet. Aufgrund dieser Variation in der Rate, mit der einzelne HLA-Allele nachgewiesen werden, wurden Versuche unternommen, Allele an jedem exprimierten HLA-Locus hinsichtlich ihrer Prävalenz zu kategorisieren. Das Ergebnis ist ein Katalog von häufigen und gut dokumentierten (CWD) HLA-Allelen und ein Katalog von seltenen und sehr seltenen HLA-Allelen.
Häufige HLA-Allele sind definiert als mit einer Häufigkeit von mindestens 0,001 in Referenzpopulationen von mindestens 1500 Individuen beobachtet. Gut dokumentierte HLA-Allele wurden ursprünglich so definiert, dass sie bei nicht verwandten Personen mindestens dreimal gemeldet wurden, und werden jetzt als mindestens fünfmal bei nicht verwandten Personen durch die Anwendung einer sequenzbasierten Typisierungsmethode (SBT) nachgewiesen, oder mindestens dreimal über eine SBT-Methode und in einem bestimmten Haplotyp bei nicht verwandten Personen. Seltene Allele sind definiert als solche, die ein- bis viermal gemeldet wurden, und sehr seltene Allele als solche, die nur einmal gemeldet wurden.
Tabelle der HLA-Allele in jeder Prävalenzkategorie
Während die aktuellen CWD- und seltenen oder sehr seltenen Bezeichnungen unter Verwendung verschiedener Datensätze und verschiedener Versionen der IMGT/HLA-Datenbank entwickelt wurden , wird der ungefähre Anteil der Allele an jedem HLA-Locus in jeder Kategorie unten gezeigt.
| HLA- Lokus |
Nr. gemeinsame Allele |
% häufige Allele |
Nr. gut dokumentierte Allele |
% gut dokumentierte Allele |
Nr. seltene Allele |
% seltene Allele |
Nr. sehr seltene Allele |
% sehr seltene Allele |
% Allele kategorisiert |
|---|---|---|---|---|---|---|---|---|---|
| EIN | 68 | 3,4% | 178 | 8,8% | 145 | 21,5% | 280 | 41,6% | ~75% |
| B | 125 | 4,8% | 242 | 9,3% | 190 | 17,6% | 468 | 43,5% | ~75% |
| C | 44 | 2,8% | 102 | 6,6% | 77 | 21,4% | 154 | 42,8% | ~74 % |
| DRB1 | 79 | 6,8% | 147 | 12,7% | 133 | 22,7% | 206 | 35,2% | ~77 % |
| DRB3 | 5 | 8,6% | 7 | 12,1% | ~21% | ||||
| DRB4 | 6 | 40,0% | 2 | 13,3% | ~53 % | ||||
| DRB5 | 5 | 25,0% | 3 | 15,0% | ~40% | ||||
| DQA1 | fünfzehn | 31,9% | 4 | 8,5% | 9 | 26,5% | 7 | 20,6% | ~88 % |
| DQB1 | 22 | 12,5 % | 8 | 4,5% | 26 | 28,9 % | 42 | 45,2% | ~91 % |
| DPA1 | 6 | 17,6% | 0 | 0.0% | 4 | 14,8% | fünfzehn | 55,6% | ~88 % |
| DPB1 | 40 | 28,8% | 14 | 9,0% | 29 | 22,7% | 29 | 32,8% | ~90% |
| Alle Loci | 415 | 5,3% | 707 | 9,0% | 613 | 20,6% | 1214 | 40,8% | ~76 % |
Untersuchung von HLA-Typen
Serotyp- und Allelnamen
Es gibt zwei parallele Nomenklatursysteme, die auf HLA angewendet werden. Das erste und älteste System basiert auf serologischer (antikörperbasierter) Erkennung. In diesem System wurden Antigenen schließlich Buchstaben und Zahlen zugeordnet (zB HLA-B27 oder abgekürzt B27). Ein paralleles System, das eine verfeinerte Definition von Allelen ermöglichte, wurde entwickelt. In diesem System wird ein "HLA" in Verbindung mit einem Buchstaben, * und einer vier- oder mehrstelligen Zahl verwendet (z. B. HLA-B*08:01, A*68:01, A*24:02 :01N N=Null), um ein spezifisches Allel an einem gegebenen HLA- Locus zu bezeichnen . HLA-Loci können weiter in MHC-Klasse I und MHC-Klasse II (oder selten D-Locus) eingeteilt werden. Alle zwei Jahre wird eine Nomenklatur veröffentlicht, um den Forschern bei der Interpretation von Serotypen zu Allelen zu helfen.
Serotypisierung
Um ein Typisierungsreagenz herzustellen, würde Blut von Tieren oder Menschen entnommen, die Blutzellen vom Serum abgetrennt und das Serum auf seine optimale Empfindlichkeit verdünnt und zur Typisierung von Zellen von anderen Individuen oder Tieren verwendet. Somit wurde die Serotypisierung zu einer Möglichkeit, HLA-Rezeptoren und Rezeptorisoformen grob zu identifizieren. Im Laufe der Jahre wurden die Serotypisierungs-Antikörper verfeinert, da sich die Techniken zur Erhöhung der Sensitivität verbesserten und immer wieder neue Serotypisierungs-Antikörper auf den Markt kamen. Eines der Ziele der Serotypanalyse ist es, Lücken in der Analyse zu schließen. Es ist möglich, basierend auf der „Quadratwurzel“, der „Maximum-Likelihood“-Methode oder der Analyse familiärer Haplotypen Vorhersagen zu treffen, um adäquat typisierte Allele zu berücksichtigen. Diese Studien mit Serotypisierungstechniken ergaben häufig, insbesondere bei nichteuropäischen oder nordostasiatischen Populationen, viele Null- oder Leerserotypen. Dies war bis vor kurzem für den Cw-Locus besonders problematisch, und fast die Hälfte der Cw-Serotypen wurde bei der Erhebung der menschlichen Bevölkerung von 1991 nicht typisiert.
Es gibt verschiedene Arten von Serotypen. Ein breiter Antigen-Serotyp ist ein grobes Maß für die Identität von Zellen. Zum Beispiel erkennt der HLA A9-Serotyp Zellen von A23- und A24-tragenden Individuen. Es kann auch Zellen erkennen, die A23 und A24 aufgrund kleiner Variationen verfehlen. A23 und A24 sind gespaltene Antigene, aber Antikörper, die für beide spezifisch sind, werden typischerweise häufiger verwendet als Antikörper gegen breite Antigene.
Handy-Typisierung
Ein repräsentativer zellulärer Assay ist die gemischte Lymphozytenkultur (MLC) und wird verwendet, um die HLA-Klasse-II-Typen zu bestimmen. Der zelluläre Assay ist beim Nachweis von HLA-Unterschieden empfindlicher als die Serotypisierung. Dies liegt daran, dass geringfügige Unterschiede, die von Alloantiseren nicht erkannt werden, T-Zellen stimulieren können. Diese Typisierung wird als Dw-Typen bezeichnet. Serotypisierter DR1 ist zellulär entweder als Dw1 oder als Dw20 usw. für andere serotypisierte DRs definiert. Tabelle zeigt assoziierte zelluläre Spezifitäten für DR-Allele. Die zelluläre Typisierung weist jedoch eine Inkonsistenz in der Reaktion zwischen Individuen des zellulären Typs auf, die manchmal anders als vorhergesagt resultieren. Zusammen mit der Schwierigkeit des zellulären Assays bei der Erzeugung und Aufrechterhaltung von zellulären Typisierungsreagenzien wird der zelluläre Assay durch ein DNA-basiertes Typisierungsverfahren ersetzt.
Gensequenzierung
Bei den Genprodukten von Allelen einer Serotypgruppe sind geringfügige Reaktionen auf Subregionen zu beobachten, die Ähnlichkeiten mit anderen Typen aufweisen. Die Sequenz der Antigene bestimmt die Antikörperreaktivitäten, so dass eine gute Sequenzierfähigkeit (oder sequenzbasierte Typisierung) die Notwendigkeit von serologischen Reaktionen überflüssig macht. Daher können verschiedene Serotypreaktionen auf die Notwendigkeit hinweisen, das HLA einer Person zu sequenzieren, um eine neue Gensequenz zu bestimmen.
Breite Antigentypen sind immer noch nützlich, wie zum Beispiel die Typisierung sehr unterschiedlicher Populationen mit vielen nicht identifizierten HLA-Allelen (Afrika, Arabien, Südostiran und Pakistan, Indien). Afrika, Südiran und Arabien zeigen die Schwierigkeit, früher besiedelte Gebiete zu tippen. Die allelische Diversität macht es notwendig, eine breite Antigentypisierung gefolgt von einer Gensequenzierung zu verwenden, da ein erhöhtes Risiko einer Fehlidentifizierung durch Serotypisierungstechniken besteht.
Am Ende entscheidet ein Workshop, basierend auf der Sequenz, entweder durch Sequenz oder durch Reaktivität, welches neue Allel in welche Serogruppe eindringt. Sobald die Sequenz verifiziert ist, wird ihr eine Nummer zugewiesen. Zum Beispiel kann ein neues Allel von B44 einen Serotyp (dh B44) und eine Allel-ID, dh B*44:65, erhalten, da es das 65. entdeckte B44-Allel ist. Marsh et al. (2005) kann als Codebuch für HLA-Serotypen und -Genotypen angesehen werden, und als ein neues Buch, das alle zwei Jahre mit monatlichen Updates in Tissue Antigens erscheint .
Phänotypisierung
Die Gentypisierung unterscheidet sich von der Gensequenzierung und Serotypisierung. Bei dieser Strategie werden PCR-Primer verwendet, die für eine varianten DNA-Region spezifisch sind (als SSP-PCR bezeichnet ). Wenn ein Produkt der richtigen Größe gefunden wird, wird davon ausgegangen, dass das HLA-Allel identifiziert wurde. Neue Gensequenzen führen oft zu einem zunehmenden Auftreten von Mehrdeutigkeit. Da die Gentypisierung auf der SSP-PCR basiert, können neue Varianten, insbesondere in den Klasse-I- und DRB1-Loci, übersehen werden.
Beispielsweise wird die SSP-PCR in der klinischen Situation häufig zur Identifizierung von HLA-Phänotypen verwendet. Ein Beispiel für einen erweiterten Phänotyp für eine Person könnte sein:
A *01:01 / *03:01 , C *07:01 / *07:02 , B *07:02 / *08:01 , DRB1 *03:01 / *15:01 , DQA1 *05:01 / *01:02 , DQB1 *02:01 / *06:02
Im Allgemeinen ist dies identisch mit dem erweiterten Serotyp: A1,A3,B7,B8,DR3,DR15(2), DQ2,DQ6(1)
Für viele Populationen, wie beispielsweise die japanische oder europäische Population, wurden so viele Patienten typisiert, dass neue Allele relativ selten sind, und daher ist SSP-PCR für die Allelauflösung mehr als ausreichend. Haplotypen können durch Typisierung von Familienmitgliedern in Gebieten der Welt erhalten werden, in denen SSP-PCR keine Allele erkennen kann und die Typisierung die Sequenzierung neuer Allele erfordert. Zu den Gebieten der Welt, in denen SSP-PCR oder Serotypisierung unzureichend sein können, gehören Zentralafrika, Ostafrika, Teile des südlichen Afrikas, Arabien, Südiran, Pakistan und Indien.
Haplotypen
Ein HLA-Haplotyp ist eine Reihe von HLA-"Genen" (Loci-Allele) nach Chromosomen, eines von der Mutter und eines vom Vater.
Der oben genannte Phänotyp ist einer der häufigsten in Irland und das Ergebnis von zwei gemeinsamen genetischen Haplotypen :
A *01:01 ; C *07:01 ; B *08:01 ; DRB1 *03:01 ; DQA1 *05:01 ; DQB1 *02:01 (Durch Serotypisierung A1-Cw7-B8-DR3-DQ2 )
die als ''Super B8'' oder ''Ahnen-Haplotyp'' bezeichnet wird und
A *03:01 ; C *07:02 ; B *07:02 ; DRB1 *15:01 ; DQA1 *01:02 ; DQB1 *06:02 (Durch Serotypisierung A3-Cw7-B7-DR15-DQ6 oder die ältere Version "A3-B7-DR2-DQ1")
Diese Haplotypen können verwendet werden, um Wanderungen in der menschlichen Bevölkerung zu verfolgen, da sie oft wie ein Fingerabdruck eines Ereignisses sind, das in der Evolution stattgefunden hat. Der Super-B8-Haplotyp ist in Westirland angereichert, nimmt entlang von Gradienten von dieser Region weg ab und kommt nur in Gebieten der Welt vor, in die Westeuropäer ausgewandert sind. Der "A3-B7-DR2-DQ1" ist weiter verbreitet, von Ostasien bis nach Iberien. Der Super-B8-Haplotyp wird mit einer Reihe von ernährungsbedingten Autoimmunerkrankungen in Verbindung gebracht. Es gibt 100.000 erweiterte Haplotypen, aber nur wenige zeigen in der menschlichen Bevölkerung einen sichtbaren und nodalen Charakter.
Rolle der Allelvariation
Studien an Menschen und Tieren implizieren als Erklärung für diese Variabilität einen heterozygoten Selektionsmechanismus, der auf diese Loci wirkt. Ein vorgeschlagener Mechanismus ist die sexuelle Selektion, bei der Weibchen in der Lage sind, Männchen mit unterschiedlichem HLA im Vergleich zu ihrem eigenen Typ zu erkennen. Während die DQ- und DP-kodierenden Loci weniger Allele aufweisen, können Kombinationen von A1:B1 ein theoretisches Potenzial von 7.755 DQ- bzw. 5.270 DP-αβ-Heterodimeren erzeugen. Obwohl bei weitem nicht diese Anzahl von Isoformen in der menschlichen Bevölkerung existiert, kann jedes Individuum 4 variable DQ- und DP-Isoformen tragen, was die potenzielle Anzahl von Antigenen erhöht, die diese Rezeptoren dem Immunsystem präsentieren können.
Untersuchungen der variablen Positionen von DP, DR und DQ zeigen, dass Peptidantigen-Kontaktreste auf Klasse-II-Molekülen am häufigsten die Variationsstelle in der Proteinprimärstruktur sind. Daher sind die Klasse-II-Peptidrezeptoren durch eine Kombination aus intensiver allelischer Variation und/oder Untereinheitspaarung in der Lage, eine fast endlose Variation von Peptiden mit einer Länge von 9 Aminosäuren oder länger zu binden, wodurch sich ineinander greifende Subpopulationen vor aufkommenden oder epidemischen Krankheiten schützen. Individuen in einer Population haben häufig unterschiedliche Haplotypen, und dies führt zu vielen Kombinationen, sogar in kleinen Gruppen. Diese Vielfalt erhöht das Überleben solcher Gruppen und verhindert die Evolution von Epitopen in Krankheitserregern, die sonst vom Immunsystem abgeschirmt werden könnten.
Antikörper
HLA-Antikörper kommen typischerweise nicht natürlich vor und werden mit wenigen Ausnahmen als Ergebnis einer immunologischen Herausforderung eines Fremdmaterials, das nicht-eigene HLAs enthält, durch Bluttransfusion, Schwangerschaft (väterlich vererbte Antigene) oder Organ- oder Gewebetransplantation gebildet.
Antikörper gegen krankheitsassoziierte HLA-Haplotypen wurden zur Behandlung schwerer Autoimmunerkrankungen vorgeschlagen.
Es wurde festgestellt, dass spenderspezifische HLA-Antikörper mit Transplantatversagen bei Nieren-, Herz-, Lungen- und Lebertransplantationen assoziiert sind.
HLA-Matching für kranke Geschwister
Bei einigen Krankheiten, die eine Transplantation von hämatopoetischen Stammzellen erfordern , kann eine genetische Präimplantationsdiagnostik verwendet werden, um ein Geschwister mit passendem HLA hervorzubringen, obwohl es ethische Überlegungen gibt.
Siehe auch
Verweise
Literaturverzeichnis
- Daniel M. Davis, The Compatibility Gene , London, Penguin Books , 2014 ( ISBN 978-0-241-95675-5 ).
- Zsolt Harsanyi und Richard Hutton, Richard, Genetic Prophecy: Beyond the Double Helix , London: Granada, 1982 ( ISBN 0-246-11760-5 ).
Externe Links
- IMGT/HLA-Sequenzdatenbank des European Bioinformatics Institute
- hla.alleles.org
- HLA-Informatikgruppe beim Anthony Nolan Trust
- Britische Gesellschaft für Histokompatibilität und Immunogenetik
- Amerikanische Gesellschaft für Histokompatibilität und Immunogenetik
- Europäische Föderation für Immunogenetik
- HistoCheck HLA-Matching-Tool für die Organ- und Stammzelltransplantation
- Allelfrequenzen an variablen immunbezogenen Loci
- Human+Leukozyten+Antigene in der US National Library of Medicine Medical Subject Headings (MeSH)
- dbMHC Home, NCBIs Datenbank des Major Histocompatibility Complex
- Seltenes Allele-Projekt in der AlleleFrequencies Net Database (AFND)
- Katalog der Common and Well Documented (CWD) Allele


