Übersetzung (Biologie) - Translation (biology)
In der Molekularbiologie und Genetik ist Translation der Vorgang, bei dem Ribosomen im Zytoplasma oder im endoplasmatischen Retikulum Proteine nach dem Prozess der Transkription von DNA zu RNA im Zellkern synthetisieren . Der gesamte Vorgang wird als Genexpression bezeichnet .
Bei der Translation wird Boten-RNA (mRNA) in einem Ribosom außerhalb des Zellkerns dekodiert, um eine spezifische Aminosäurekette oder ein Polypeptid zu produzieren . Das Polypeptid faltet sich später zu einem aktiven Protein und erfüllt seine Funktionen in der Zelle. Das Ribosom erleichtert die Entschlüsselung, indem es die Bindung komplementärer tRNA- Anticodon- Sequenzen an mRNA- Codons induziert . Die tRNAs tragen spezifische Aminosäuren, die beim Durchlaufen der mRNA zu einem Polypeptid verkettet und vom Ribosom "gelesen" werden.
Die Übersetzung verläuft in drei Phasen:
- Initiation : Das Ribosom versammelt sich um die Ziel-mRNA. Die erste tRNA wird am Startcodon angehängt .
- Elongation : Die letzte von der kleinen ribosomalen Untereinheit ( Akkommodation ) validierte tRNA überträgt die von ihr getragene Aminosäure auf die große ribosomale Untereinheit , die sie an die zuvor zugelassene tRNA bindet ( Transpeptidierung ). Das Ribosom bewegt sich dann zum nächsten mRNA-Codon, um den Prozess fortzusetzen ( Translokation ), wodurch eine Aminosäurekette entsteht.
- Termination : Wenn ein Stopcodon erreicht wird, setzt das Ribosom das Polypeptid frei. Der ribosomale Komplex bleibt intakt und geht zur nächsten zu translatierenden mRNA über.
Bei Prokaryoten (Bakterien und Archaeen) erfolgt die Translation im Zytosol, wo die großen und kleinen Untereinheiten des Ribosoms an die mRNA binden. Bei Eukaryoten erfolgt die Translation im Zytoplasma oder durch die Membran des endoplasmatischen Retikulums in einem Prozess, der als kotranslationale Translokation bezeichnet wird . Bei der kotranslationalen Translokation bindet der gesamte Ribosom/mRNA-Komplex an die äußere Membran des rauen endoplasmatischen Retikulums (ER) und das neue Protein wird synthetisiert und in das ER freigesetzt; das neu erzeugte Polypeptid kann im ER für den zukünftigen Vesikeltransport und die Sekretion außerhalb der Zelle gespeichert oder sofort sekretiert werden.
Viele Typen transkribierter RNA, wie Transfer-RNA, ribosomale RNA und kleine nukleäre RNA, werden nicht in Proteine übersetzt.
Eine Reihe von Antibiotika wirken durch Hemmung der Translation. Dazu gehören Anisomycin , Cycloheximid , Chloramphenicol , Tetracyclin , Streptomycin , Erythromycin und Puromycin . Prokaryontische Ribosomen haben eine andere Struktur als eukaryontische Ribosomen, und daher können Antibiotika spezifisch bakterielle Infektionen bekämpfen, ohne die Zellen eines eukaryontischen Wirts zu schädigen .
Grundmechanismen

Der grundlegende Prozess der Proteinproduktion ist die Zugabe einer Aminosäure nach der anderen an das Ende eines Proteins. Diese Operation wird von einem Ribosom durchgeführt . Ein Ribosom besteht aus zwei Untereinheiten, einer kleinen Untereinheit und einer großen Untereinheit. Diese Untereinheiten kommen vor der Translation von mRNA in ein Protein zusammen, um einen Ort für die durchzuführende Translation und die Herstellung eines Polypeptids bereitzustellen. Die Wahl des hinzuzufügenden Aminosäuretyps wird durch ein mRNA- Molekül bestimmt. Jede hinzugefügte Aminosäure stimmt mit einer Subsequenz aus drei Nukleotiden der mRNA überein. Für jedes dieser möglichen Tripletts wird die entsprechende Aminosäure akzeptiert. Die aufeinanderfolgenden Aminosäuren, die der Kette hinzugefügt werden, werden mit aufeinanderfolgenden Nukleotidtripletts in der mRNA abgeglichen. Auf diese Weise bestimmt die Nukleotidsequenz in der Matrizen-mRNA-Kette die Aminosäuresequenz in der erzeugten Aminosäurekette. Die Addition einer Aminosäure erfolgt am N-Terminus des Peptids und daher wird die Translation als Carboxyl-zu-Amino-gerichtet bezeichnet.
Die mRNA trägt als Ribonukleotidsequenz kodierte genetische Information von den Chromosomen zu den Ribosomen. Die Ribonukleotide werden von einer Translationsmaschinerie in einer Sequenz von Nukleotidtripletts, die Codons genannt werden, "gelesen" . Jedes dieser Tripletts kodiert für eine bestimmte Aminosäure .
Die Ribosomenmoleküle übersetzen diesen Code in eine bestimmte Aminosäuresequenz. Das Ribosom ist eine Struktur aus mehreren Untereinheiten, die rRNA und Proteine enthält. Es ist die "Fabrik", in der Aminosäuren zu Proteinen zusammengesetzt werden. tRNAs sind kleine nichtkodierende RNA-Ketten (74–93 Nukleotide), die Aminosäuren zum Ribosom transportieren. tRNAs haben eine Stelle für die Anheftung von Aminosäuren und eine Stelle, die als Anticodon bezeichnet wird. Das Anticodon ist ein zu dem mRNA-Triplett komplementäres RNA-Triplett, das für ihre Cargo- Aminosäure kodiert .
Aminoacyl-tRNA-Synthetasen ( Enzyme ) katalysieren die Bindung zwischen spezifischen tRNAs und den Aminosäuren, die ihre Anticodon-Sequenzen erfordern . Das Produkt dieser Reaktion ist eine Aminoacyl-tRNA . In Bakterien wird diese Aminoacyl-tRNA von EF-Tu zum Ribosom transportiert , wo mRNA-Codons durch komplementäre Basenpaarung an spezifische tRNA- Anticodons angepasst werden . Aminoacyl-tRNA-Synthetasen, die tRNAs mit den falschen Aminosäuren fehlpaaren, können fehlgeladene Aminoacyl-tRNAs produzieren, was zu unpassenden Aminosäuren an der entsprechenden Position im Protein führen kann. Diese "Fehlübersetzung" des genetischen Codes tritt in den meisten Organismen natürlich in geringen Mengen auf, aber bestimmte zelluläre Umgebungen verursachen eine Zunahme der permissiven mRNA-Decodierung, manchmal zum Vorteil der Zelle.
Das Ribosom hat zwei Bindungsstellen für tRNA. Sie sind die Aminoacylstelle (abgekürzt A), die Peptidylstelle/Ausgangsstelle (abgekürzt P/E). In Bezug auf die mRNA sind die drei Stellen von 5' zu 3' EPA orientiert, da sich Ribosomen zum 3'-Ende der mRNA bewegen. Die A-Stelle bindet die ankommende tRNA mit dem komplementären Codon auf der mRNA. Die P/E-Stelle hält die tRNA mit der wachsenden Polypeptidkette. Wenn eine Aminoacyl-tRNA anfänglich an ihr entsprechendes Codon auf der mRNA bindet, befindet sie sich an der A-Stelle. Dann bildet sich eine Peptidbindung zwischen der Aminosäure der tRNA an der A-Stelle und der Aminosäure der geladenen tRNA an der P/E-Stelle. Die wachsende Polypeptidkette wird auf die tRNA in der A-Stelle übertragen. Es erfolgt eine Translokation, wobei die tRNA in die P/E-Stelle verschoben wird, jetzt ohne Aminosäure; die tRNA, die sich an der A-Stelle befand und nun mit der Polypeptidkette beladen ist, wird an die P/E-Stelle verschoben und die tRNA verlässt und eine andere Aminoacyl-tRNA tritt in die A-Stelle ein, um den Vorgang zu wiederholen.
Nachdem die neue Aminosäure an die Kette hinzugefügt wurde und die tRNA aus dem Ribosom und in das Zytosol freigesetzt wurde, wird die Energie durch die Hydrolyse eines an die Translocase EF-G (in Bakterien ) und a/eEF . gebundenen GTP -2 (in Eukaryoten und Archaeen ) bewegt das Ribosom ein Codon nach unten zum 3'-Ende . Die Energie, die für die Translation von Proteinen benötigt wird, ist signifikant. Für ein Protein, das n Aminosäuren enthält, beträgt die Anzahl der für die Translation erforderlichen hochenergetischen Phosphatbindungen 4 n -1. Die Übersetzungsrate variiert; er ist in prokaryontischen Zellen signifikant höher (bis zu 17–21 Aminosäurereste pro Sekunde) als in eukaryontischen Zellen (bis zu 6–9 Aminosäurereste pro Sekunde).
Obwohl die Ribosomen normalerweise als genaue und prozessive Maschinen gelten, unterliegt der Übersetzungsprozess Fehlern, die entweder zur Synthese fehlerhafter Proteine oder zum vorzeitigen Abbruch der Übersetzung führen können. Die Fehlerrate bei der Synthese von Proteinen wurde auf zwischen 1/10 5 und 1/10 3 falsch eingebaute Aminosäuren geschätzt , abhängig von den experimentellen Bedingungen. Die Rate des vorzeitigen Abbruchs der Translation wurde stattdessen auf eine Größenordnung von 10 –4 Ereignissen pro translatiertem Codon geschätzt . Die richtige Aminosäure wird durch Aminoacyltransferasen kovalent an die richtige Transfer-RNA (tRNA) gebunden . Die Aminosäure ist mit ihrer Carboxylgruppe über eine Esterbindung mit dem 3'-OH der tRNA verbunden . Wenn an die tRNA eine Aminosäure gebunden ist, wird die tRNA als "geladen" bezeichnet. Die Initiation beinhaltet die Bindung der kleinen Untereinheit des Ribosoms an das 5'-Ende der mRNA mit Hilfe von Initiationsfaktoren (IF). Bei Bakterien und einer Minderheit von Archaeen beinhaltet die Initiation der Proteinsynthese die Erkennung einer purinreichen Initiationssequenz auf der mRNA, die als Shine-Delgarno-Sequenz bezeichnet wird. Die Shine-Delgarno-Sequenz bindet an eine komplementäre pyrimidinreiche Sequenz am 3'-Ende des 16S-rRNA-Teils der 30S-ribosomalen Untereinheit. Die Bindung dieser komplementären Sequenzen stellt sicher, dass die ribosomale 30S-Untereinheit an die mRNA gebunden und so ausgerichtet ist, dass das Initiationscodon im 30S-Teil der P-Stelle platziert ist. Sobald die mRNA und die 30S-Untereinheit richtig gebunden sind, bringt ein Initiationsfaktor den Initiator-tRNA-Aminosäure-Komplex, f-Met-tRNA, an die 30S P-Stelle. Die Initiationsphase ist abgeschlossen, sobald sich eine 50S-Untereinheit an die 30-Untereinheit anschließt und ein aktives 70S-Ribosom bildet. Die Terminierung des Polypeptids tritt auf, wenn die A-Stelle des Ribosoms von einem Stoppcodon (UAA, UAG oder UGA) auf der mRNA besetzt ist. tRNA kann normalerweise Stoppcodons nicht erkennen oder daran binden. Stattdessen induziert das Stoppcodon die Bindung eines Freisetzungsfaktorproteins . (RF1 & RF2), der die Zerlegung des gesamten Ribosom/mRNA-Komplexes durch Hydrolyse der Polypeptidkette vom Peptidyltransferasezentrum des Ribosoms auslöst Medikamente oder spezielle Sequenzmotive auf der mRNA können die ribosomale Struktur so verändern, dass nahe verwandte tRNAs werden an das Stoppcodon statt an die Freisetzungsfaktoren gebunden. In solchen Fällen des "translationalen Durchlesens" wird die Translation fortgesetzt, bis das Ribosom auf das nächste Stoppcodon trifft.
Der Translationsprozess ist sowohl in eukaryontischen als auch in prokaryontischen Organismen stark reguliert. Die Regulation der Translation kann die globale Geschwindigkeit der Proteinsynthese beeinflussen, die eng mit dem metabolischen und proliferativen Zustand einer Zelle verbunden ist. Darüber hinaus haben neuere Arbeiten gezeigt, dass auch genetische Unterschiede und deren nachfolgende Expression als mRNAs die Translationsrate RNA-spezifisch beeinflussen können.
Klinische Bedeutung
Die translationale Kontrolle ist entscheidend für die Entwicklung und das Überleben von Krebs . Krebszellen müssen häufig die Translationsphase der Genexpression regulieren, obwohl nicht vollständig verstanden wird, warum die Translation über Schritte wie die Transkription abzielt. Während Krebszellen oft genetisch veränderte Translationsfaktoren aufweisen, ist es für Krebszellen viel häufiger, dass sie das Niveau vorhandener Translationsfaktoren modifizieren. Mehrere wichtige onkogene Signalwege, darunter der RAS-MAPK- , PI3K/AKT/mTOR- , MYC- und WNT-β-Catenin- Weg, programmieren das Genom schließlich durch Translation neu. Krebszellen kontrollieren auch die Translation, um sich an zellulären Stress anzupassen. Bei Stress übersetzt die Zelle mRNAs, die den Stress mildern und das Überleben fördern können. Ein Beispiel hierfür ist die Expression von AMPK bei verschiedenen Krebsarten; seine Aktivierung löst eine Kaskade aus, die es dem Krebs letztendlich ermöglichen kann, der Apoptose (programmiertem Zelltod) zu entkommen, die durch Nahrungsentzug ausgelöst wird. Zukünftige Krebstherapien können eine Unterbrechung der Translationsmaschinerie der Zelle beinhalten, um den nachgelagerten Auswirkungen von Krebs entgegenzuwirken.
Mathematische Modellierung der Übersetzung
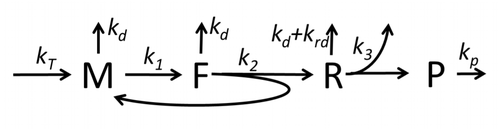
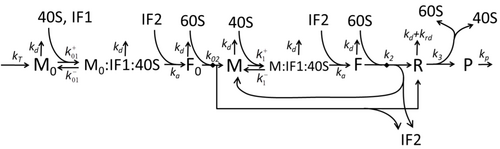
Die Beschreibung des Transkriptions-Übersetzungs-Prozesses, die nur die grundlegendsten „elementaren“ Prozesse erwähnt, besteht aus:
- Herstellung von mRNA-Molekülen (einschließlich Spleißen),
- Initiation dieser Moleküle mit Hilfe von Initiationsfaktoren (z. B. kann die Initiation den Zirkularisierungsschritt umfassen, obwohl dies nicht allgemein erforderlich ist),
- Einleitung der Translation, Rekrutierung der kleinen ribosomalen Untereinheit,
- Zusammenbau von vollständigen Ribosomen,
- Elongation (dh Bewegung von Ribosomen entlang der mRNA unter Produktion von Protein),
- Beendigung der Übersetzung,
- Abbau von mRNA-Molekülen,
- Abbau von Proteinen.
Der Prozess des Aminosäureaufbaus zur Erzeugung von Proteinen in der Translation ist seit langem Gegenstand verschiedener physikalischer Modelle, beginnend mit ersten detaillierten kinetischen Modellen wie ua unter Berücksichtigung stochastischer Aspekte der Translation und unter Verwendung von Computersimulationen. In den letzten vier Jahrzehnten wurden viele auf der chemischen Kinetik basierende Modelle der Proteinsynthese entwickelt und analysiert. Über die chemische Kinetik hinaus wurden verschiedene Modellierungsformalismen wie Totally Asymmetric Simple Exclusion Process (TASEP) , Probabilistic Boolean Networks (PBN) , Petrinetze und Max-Plus-Algebra angewendet, um die detaillierte Kinetik der Proteinsynthese oder einiger ihrer Stufen zu modellieren. Nach dem Paradigma „nützliche Modelle sind einfach und erweiterbar“ wurde ein Grundmodell der Proteinsynthese entwickelt, das alle acht „elementaren“ Prozesse berücksichtigt . Das einfachste Modell M0 wird durch den reaktionskinetischen Mechanismus repräsentiert (Abbildung M0). Es wurde verallgemeinert, um die Bindung von 40S, 60S und Initiationsfaktoren (IF) einzuschließen (Abbildung M1'). Es wurde weiter erweitert, um die Wirkung von microRNA auf die Proteinsynthese einzubeziehen. Die meisten Modelle in dieser Hierarchie können analytisch gelöst werden. Diese Lösungen wurden verwendet, um „kinetische Signaturen“ verschiedener spezifischer Mechanismen der Syntheseregulation zu extrahieren.
Genetischer Code
Während andere Aspekte wie die 3D-Struktur, Tertiärstruktur genannt , von Proteinen nur mit ausgeklügelten Algorithmen vorhergesagt werden können , kann die Aminosäuresequenz, Primärstruktur genannt , allein aus der Nukleinsäuresequenz mit Hilfe einer Translationstabelle bestimmt werden .
Dieser Ansatz liefert möglicherweise nicht die korrekte Aminosäurezusammensetzung des Proteins, insbesondere wenn unkonventionelle Aminosäuren wie Selenocystein in das Protein eingebaut werden, das durch ein konventionelles Stoppcodon in Kombination mit einer nachgeschalteten Haarnadel (SElenoCysteine Insertion Sequence, oder .) kodiert wird SECIS).
Es gibt viele Computerprogramme, die eine DNA/RNA-Sequenz in eine Proteinsequenz übersetzen können. Normalerweise wird dies unter Verwendung des genetischen Standardcodes durchgeführt, jedoch können nur wenige Programme alle "speziellen" Fälle behandeln, wie die Verwendung der alternativen Initiationscodons, die biologisch bedeutsam sind. Zum Beispiel kodiert das seltene alternative Startcodon CTG für Methionin, wenn es als Startcodon verwendet wird, und für Leucin in allen anderen Positionen.
Beispiel: Kondensierte Übersetzungstabelle für den genetischen Standardcode (von der NCBI-Taxonomie-Webseite ).
AAs = FFLLSSSSYY**CC*WLLLLPPPPHHQQRRRRIIIMTTTTNNKKSSRRVVVVAAAADDEEGGGG Starts = ---M---------------M---------------M---------------------------- Base1 = TTTTTTTTTTTTTTTTCCCCCCCCCCCCCCCCAAAAAAAAAAAAAAAAGGGGGGGGGGGGGGGG Base2 = TTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGG Base3 = TCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAG
Die Zeile "Starts" zeigt drei Startcodons an, UUG, CUG und das sehr häufige AUG. Es zeigt auch den ersten Aminosäurerest an, wenn es als Start interpretiert wird: In diesem Fall ist es alles Methionin.
Übersetzungstabellen
Auch bei der Arbeit mit gewöhnlichen eukaryontischen Sequenzen wie dem Hefegenom ist es oft erwünscht, alternative Übersetzungstabellen verwenden zu können – nämlich für die Translation der mitochondrialen Gene. Derzeit werden die folgenden Übersetzungstabellen von der NCBI Taxonomy Group für die Übersetzung der Sequenzen in GenBank definiert :
- Der Standardcode
- Der mitochondriale Code von Wirbeltieren
- Der mitochondriale Code der Hefe
- Der mitochondriale Code von Schimmel, Protozoen und Coelenterat sowie der Mycoplasma/Spiroplasma-Code
- Der mitochondriale Code von Wirbellosen
- Der Ciliat-, Dasycladacean- und Hexamita-Nuklearcode
- Der Kinetoplast-Code
- Der mitochondriale Code von Stachelhäutern und Plattwürmern
- Der euplotide Nuklearcode
- Der bakterielle, archaische und pflanzliche Plastidencode
- Der alternative Hefe-Nuklearcode
- Der mitochondriale Code der Ascidien
- Der alternative mitochondriale Code des Plattwurms
- Der nukleare Code von Blepharisma
- Der mitochondriale Code der Chlorophycea
- Der mitochondriale Code der Trematoden
- Der mitochondriale Code von Scenedesmus obliquus
- Der mitochondriale Code von Thraustochytrium
- Der mitochondriale Code von Pterobranchia
- Die Kandidatenteilung SR1 und der Gracilibakterien-Code
- Die Pachysolen tannophilus Kern Code
- Der karyoreliktische Nuklearcode
- Der Nuklearcode von Condylostoma
- Der Kerncode von Mesodinium
- Der peritriche Nuklearcode
- Der nukleare Code von Blastocrithidia
- Der mitochondriale Code von Cephalodiscidae
Siehe auch
Verweise
Weiterlesen
- Champe PC, Harvey RA, Ferrier DR (2004). Lippincotts illustrierte Rezensionen: Biochemie (3. Aufl.). Hagerstwon, MD: Lippincott Williams & Wilkins. ISBN 0-7817-2265-9.
- Cox M, Nelson DR, Lehninger AL (2005). Lehninger Prinzipien der Biochemie (4. Aufl.). San Francisco...: WH Freeman. ISBN 0-7167-4339-6.
- Malys N, McCarthy JE (März 2011). "Übersetzungsinitiation: Variationen im Mechanismus können erwartet werden". Zelluläre und molekulare Biowissenschaften . 68 (6): 991–1003. doi : 10.1007/s00018-010-0588-z . PMID 21076851 . S2CID 31720000 .