Alternative Verlängerung von Telomeren - Alternative Lengthening of Telomeres
Die alternative Verlängerung von Telomeren (auch als " ALT " bekannt) ist ein Telomerase- unabhängiger Mechanismus, durch den Krebszellen den Abbau von Telomeren vermeiden .
Hintergrund
An jedem Ende der Chromosomen der meisten eukaryotischen Zellen befindet sich ein Telomer: eine Region repetitiver Nukleotidsequenzen , die das Ende des Chromosoms vor einer Verschlechterung oder vor einer Fusion mit benachbarten Chromosomen schützt. Bei jeder Zellteilung werden die Telomere kürzer, was schließlich eine weitere Zellteilung verhindert. Gesunde adulte somatische Zellen in Säugetieren haben keine aktiven Telomeraseenzyme, so dass Krebszellen aufhören, sich zu vermehren, es sei denn, sie haben eine Mutation, die die Telomere wiederherstellt. Dies ist häufig darauf zurückzuführen, dass ein Telomeraseenzym reaktiviert wird, es treten jedoch auch alternative Mechanismen auf.
Mechanismus der rekombinationsvermittelten Telomersynthese
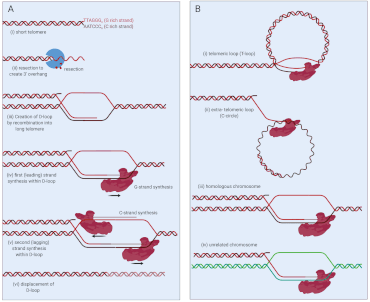
Der wichtigste alternative Verlängerungsmechanismus für Telomere ist eine Art homologer Rekombination, die als Break-induzierte Telomersynthese (BITS) bezeichnet wird. Normalerweise ermöglicht die homologe Rekombination die Reparatur gebrochener DNA-Stränge durch Aneinanderreihung mit einer passenden Sequenz unbeschädigter DNA. Bei BITS wird dieser Mechanismus jedoch zur Verlängerung von Telomeren verwendet. Da sich Telomere von Natur aus wiederholen, sind passende Sequenzen weit verbreitet.
In vorgeschlagenen Modellen für die Funktionsweise von BITS beginnt der Prozess mit der Resektion eines beschädigten Telomerendes: Einer der Stränge wird weggeschnitten, um einen einzelnen DNA-Strang (den Guanosin- reichen Strang) bereitzustellen , an den ein passender (homologer) Strang gebunden werden kann ) Schablone, die eine sogenannte Verschiebungsschleife ( D-Schleife ) bildet (Abbildung 1a). In der ALT gibt es Hinweise darauf, dass dieses Template besteht aus: (i) einer zentromeren proximalen Sequenz desselben Chromosoms ( T-Schleife ), (ii) kreisförmigen extrachromosomalen Telomersequenzen (C-Kreise), (iii) homologen Chromosomen oder iv) andere Chromosomen (Abbildung 1b). ALT kann aus einer Kombination einiger oder aller dieser Vorlagen entstehen. Da sich Telomere stark wiederholen, ist die Invasion zwischen oder innerhalb von Telomeren nicht durch das Erfordernis einer erweiterten Homologie bei der homologen Rekombination beschränkt. Nach der Bildung der D-Schleife verlängert die DNA-Polymerase δ das eingedrungene G-Strang-Ende und kopiert das Material über den ursprünglichen Bruchpunkt hinaus, was zur Initiierung der Synthese des C-Strangs mit verzögertem Strang führt, ebenfalls durch DNA-Polymerase δ.
Das zweite Merkmal von ALT ist die Herstellung eines nicht konservativen DNA-Produkts am Telomer. Am Ende der Kopierreaktion enthalten beide Stränge völlig neue DNA. Dies unterscheidet sich von der normalen "halbkonservativen" DNA-Replikation , bei der ein Strang neu synthetisiert wird und der andere von der ursprünglichen Matrize stammt. Auf diese Weise ermöglicht ALT das Kopieren ganzer Telomersequenzen von einem Chromosom auf ein anderes, ohne die Länge oder Integrität der kopierten Sequenz zu beeinflussen. Neuere Arbeiten legen nahe, dass das Kopieren von ALT-DNA (BITS) über ein D-Loop- Migrationsmodell erfolgt, das durch die Beobachtung nichtkonservativer und nicht halbkonservativer Produkte der bruchinduzierten Replikation an ALT-Telomeren und D-Loop-förmig unterstützt wird Produkte, die bei der zweidimensionalen Gelelektrophorese an Stellen beobachtet wurden, an denen BIR durchgeführt wurde.
Verweise
-
^ a b
O'Rourke, JJ; Bythell-Douglas, R; Dunn, EA; Deans, AJ (Dezember 2019). "ALT-Kontrolle, streichen: FANCM als Anti-Krebs-Ziel bei der alternativen Verlängerung von Telomeren" . Nucleus (Austin, Tex.) . 10 (1): 221–230. doi : 10.1080 / 19491034.2019.1685246 . PMC 6949022 . PMID 31663812 .
 Text wurde aus dieser Quelle kopiert, die unter einer Creative Commons Attribution 4.0 International License verfügbar ist .
Text wurde aus dieser Quelle kopiert, die unter einer Creative Commons Attribution 4.0 International License verfügbar ist .
- ^ Zhang, JM; Yadav, T; Ouyang, J; Lan, L; Zou, L (22. Januar 2019). "Alternative Verlängerung von Telomeren durch zwei unterschiedliche bruchinduzierte Replikationswege" . Zellenberichte . 26 (4): 955–968.e3. doi : 10.1016 / j.celrep.2018.12.102 . PMC 6366628 . PMID 30673617 .
- ^ O'Rourke, Julienne J; Bythell-Douglas, Rohan; Dunn, Elyse A; Dekane, Andrew J (1. Januar 2019). "ALT-Kontrolle, streichen: FANCM als Anti-Krebs-Ziel bei der alternativen Verlängerung von Telomeren" . Kern . 10 (1): 221–230. doi : 10.1080 / 19491034.2019.1685246 . PMC 6949022 . PMID 31663812 .
- ^ Donnianni, RA; Zhou, ZX; Lujan, SA; Al-Zain, A; Garcia, V; Glancy, E; Burkholder, AB; Kunkel, TA; Symington, LS (7. November 2019). "DNA-Polymerase-Delta synthetisiert beide Stränge während der bruchinduzierten Replikation" . Molekulare Zelle . 76 (3): 371–381.e4. doi : 10.1016 / j.molcel.2019.07.033 . PMC 6862718 . PMID 31495565 .
- ^ Min, J; Wright, WIR; Shay, JW (15. Oktober 2017). "Alternative Verlängerung von Telomeren, die durch mitotische DNA-Synthese vermittelt werden, führt zu bruchinduzierten Replikationsprozessen" . Molekular- und Zellbiologie . 37 (20). doi : 10.1128 / MCB.00226-17 . PMC 5615184 . PMID 28760773 .
- ^ Sneeden, JL; Grossi, SM; Tappin, ich; Hurwitz, J; Heyer, WD (Mai 2013). "Rekonstitution der Rekombinations-assoziierten DNA-Synthese mit menschlichen Proteinen" . Nukleinsäureforschung . 41 (9): 4913–25. doi : 10.1093 / nar / gkt192 . PMC 3643601 . PMID 23535143 .
