Interne Ribosomen-Eintrittsstelle (IRES) des Hepatitis-A-Virus - Hepatitis A virus internal ribosome entry site (IRES)
| Interne Ribosomen-Eintrittsstelle (IRES) des Hepatitis-A-Virus | |
|---|---|
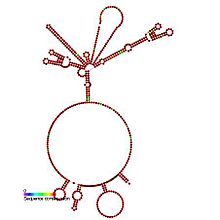 Vorhergesagte Sekundärstruktur und Sequenzkonservierung von IRES_HepA
| |
| Bezeichner | |
| Symbol | IRES_HepA |
| Alt. Symbole | HepA_IRES |
| Rfam | RF00228 |
| Andere Daten | |
| RNA- Typ | Cis-Reg. ; IRES |
| Domain(s) | Viren |
| GEHEN | GO:0043022 |
| SO | SO:0000243 |
| PDB- Strukturen | PDBe |
Diese Familie repräsentiert die interne Ribosomen-Eintrittsstelle (IRES) des Hepatitis-A-Virus . HAV IRES ist eine 450 Nukleotide lange Sequenz, die sich in der 735 nt langen 5'-UTR (untranslatierten Region) des viralen RNA-Genoms von Hepatitis A befindet . IRES-Elemente ermöglichen eine Cap- und End-unabhängige Translation von mRNA in der Wirtszelle. Das IRES erreicht dies, indem es die interne Initiation der Translation vermittelt, indem es einen ribosomalen 40S-Prä-Initiationskomplex direkt an das Initiationscodon rekrutiert und die Notwendigkeit des eukaryotischen Initiationsfaktors eIF4F eliminiert.
Geschichte und Hintergrund
Das IRES wurde erstmals 1988 von Norman Sonenberg im RNA-Genom von Picornaviridae entdeckt . Ursprünglich wurde die identifizierte 5'-UTR des Poliovirus (PV), die die interne Initiation für die Proteinsynthese steuerte, als Ribosomenlandeplatz (RLP) bezeichnet. Dieser Begriff wurde kurz darauf durch die interne Ribosomeneintrittsstelle ersetzt, die heute auch häufiger verwendet wird. 1988 wurden nur das Enzephalomykoarditis-Virus ( EMCV ) und PV verwendet, um die IRES-Fähigkeit nachzuweisen. Die Forschung zur Identifizierung des IRES in HAV wurde 1993 von Michael J. Glass, Xi-Yu Jia und Donald F. Summers durchgeführt. Ihre Forschung zeigte, dass das IRES von HAV stromabwärts von Nukleotid 45 lokalisiert war und bis zu Nukleotid 734 umfasste. Die Cap-unabhängige interne Initiation der Proteinsynthese unterscheidet sich von der normalen zellulären cap-abhängigen Translationsinitiation. eIF4 (eukaryotischer Initiationsfaktor 4) als Gesamtkomplex bestehend aus eIF4A, eIF4B, eIF4E und eIF4G. eIF4F wird verwendet, um sich auf den Komplex zu beziehen, der eIF4E, 4G und 4A umfasst. Bei cap-abhängiger Translationsinitiation bindet eIF4F an das m7G-Cap am 5'-UTR-Ende, rekrutiert die kleine ribosomale 40S- Untereinheit und scannt stromabwärts nach dem AUG-Startcodon. Es hat sich gezeigt, dass eine Cap-unabhängige interne Initiation entweder mit Teilen von eIF4 oder dem gesamten Komplex interferiert.
Funktion
Unbeschichtet im Zytoplasma initiiert das HAV-Genom die Translation unabhängig von einer 5'-Kappe, um seine viralen Proteine zu synthetisieren. Die Sekundärstruktur des HAV-IRES ist sowohl notwendig als auch ausreichend, damit das Genom ein Ribosom rekrutieren und die Translation einleiten kann. Ein Ribosom der Wirtszelle erkennt das IRES und tritt direkt an der Sequenz ein, anstatt vom 5'-Ende zu scannen. Das HAV-Genom kodiert nicht für Proteine, die über die Fähigkeit zum Abschalten von Wirtsproteinen verfügen. Daher muss die HAV-IRES mit der m7G-gekappten mRNA der Wirtszelle konkurrieren. Leider ist die HAV-IRES-Initiation der Translation nicht so effizient wie eine typische m7G-Cap einer Wirtszelle. Obwohl die HAV-IRES-Struktur eine Affinität für eIF4F aufweist, ist ihre Affinität nicht annähernd so hoch wie die der mit einer Kappe versehenen mRNAs der Wirtszelle. Dies führt dazu, dass ein längerer Zeitraum benötigt wird, bis die maximale Ausscheidung des Virus erreicht ist. Dies führt auch dazu, dass der Zelltod nur durch Immunantworten des Wirts und nicht durch Lyse auftritt.
Unterdrückung von IRES
Da eIF4F eine wesentliche Rolle für die Initiierung von HAV-IRES spielt, ist es ein Ziel für die Unterdrückung von IRES. Die Spaltung von eIF4G , einem Proteingerüst von eIF4F, durch sequenzspezifische Proteasen 2A-Protease oder L-Protease führt zu einer stark gehemmten HAV-IRES-Aktivität. Diese Proteasen werden von anderen Mitgliedern der Familie der Picornaviridae kodiert . Das Maul- und Klauenseuchevirus (FMDV) zum Beispiel kodiert diese Proteasen, um die zelluläre mRNA-Translation zu hemmen, während die virale RNA translatiert werden kann. Das Erfordernis eines intakten eIF4G für die IRES-Initiation ist neben anderen Picornaviren spezifisch für HAV IRES. Das eIF4E-bindende Protein I (4E-BP1) wird auch das eIF4G-Protein stören. 4E-BP1 funktioniert durch Sequestrieren von eIF4E, das dadurch seine Assoziation mit eIF4G hemmt und zu einer HAV-IRES-Inaktivierung führt (1). Ein weiteres Verfahren zur Inaktivierung der eIF4F-Aktivität erfolgt durch die Wirkung des m7GpppG-Cap-Analogons, das auf eIF4E abzielt und dann seine Assoziation mit den mit Caps versehenen 5'-Enden von mRNAs verhindern kann. Der genaue Mechanismus, in dem dieses Cap-Analogon mit IRES interferiert, ist nicht klar, aber es wird vermutet, dass die Bindung dieses Analogs an eIF4E zu einer Konformationsänderung von eIF4G führt, die die normale Funktion von eIF4G stört.
Glyceraldehyd-3-Phosphat-Dehydrogenase (GAPDH)
Glyceraldehyd-3-Phosphat-Dehydrogenase ( GAPDH ) ist ein zelluläres Enzym, das typischerweise an der Glykolyse beteiligt ist . Es ist bekannt, dass GAPDH an die überlappenden Stellen innerhalb der Stammschleife IIIa innerhalb des HAV-IRES bindet. Die Stammschleife IIIa enthält eine UU- Nukleotid-Deletion innerhalb einer 5-Nukleotid-Sequenz, die die IRES-Aktivität verstärkt. GAPDH, das effektiv an diese Region bindet, destabilisiert die Sekundärstruktur, die das IRES bildet, und unterdrückt die Fähigkeit des IRES, die cap-unabhängige Translation durchzuführen.
La-Proteinunterdrückung
Als Wirtszellprotein, das weithin und ausschließlich in eukaryontischen Zellen vorkommt, bindet das La-Protein während der mRNA-Translation sowie der RNA-Replikation direkt an spezifische Regionen auf der HAV-IRES . In einer Studie aus dem Jahr 2008 wurde beobachtet, dass zytoplasmatisches La die Initiierung von HAV-IRES reduziert. Im Jahr 2014 zeigte eine neuere Studie jedoch eine erfolgreiche Hemmung (in vivo) des La-Proteins als vorgeschlagene Methode zur Hemmung der HAV-IRES-Translation und -Replikation, was bedeutet, dass es höchstwahrscheinlich eine wesentliche Rolle bei der HAV-Translation und -Replikation spielt.
Amantadin
Amantadin , ein trizyklisches symmetrisches Amin , ist ein bewährter Suppressor, der spezifisch die HAV-IRES-abhängige Translation von HAV-RNA hemmt. Ein Experiment aus dem Jahr 2005 zeigte, dass Amantadin die HAV-IRES-Translation unterdrückt und keine Interferon-Reaktion auslöste, was auf eine vielversprechende antivirale Anwendung von Amantadin hinweist. Beim Influenza-A-Virus besteht seine primäre Wirkungsweise als antivirales Mittel darin, die Enthüllung des viralen Genoms zu verhindern, das die HAV-IRES-vermittelte Translation und Replikation hemmt. Die Wirksamkeit von Amantadin beruht auf der IRES-Position in der 5'-NTR-Region, die eine hohe Affinität für antivirale Mittel aufweist, was es zu einem wirksamen Ziel macht. Es wurde auch gezeigt, dass das M2-Protein des Influenza-A-Virus ein weiteres brauchbares Ziel für das potenzielle antivirale Mittel sein könnte.
Alle Picornaviren enthalten IRES. Es gibt vier Klassen von IRES innerhalb der Familie der Picornaviridae , die von 270 bis 450 nt reichen. Unter den Picornaviren enthalten viele 5'-UTRs auch zusätzliche Strukturelemente stromaufwärts, die das virale Genom bei der Replikation unterstützen können. Viele Picornavirus-IRES erlauben auch vielen Viren, die cap-abhängige Initiation zu blockieren, was zum Abschalten der Proteinsynthese der Wirtszelle führt. Die vier Klassen sind Entero-/Rhinovirus-IRES, Cardio-/Aphthovirus-IRES, HAV-IRES, HCV-ähnliche Picornavirus-IRES. Diese IRES werden nach ihren Nukleotidsequenzen kategorisiert, weisen jedoch strukturelle Ähnlichkeiten auf, da es die RNA-Struktur ist, die die Fähigkeit besitzt, intern eine Translationsmaschinerie zu rekrutieren. Entero-/Rhinovirus-IRES-Elemente teilen einige strukturelle Motive mit HAV-IRES. HAV-IRES, Entero-/Rhinovirus- und Cardio-/Aptovirus-IRES sind alle ungefähr 450 nt groß, unterscheiden sich jedoch stark in ihren Strukturen. Ein Kardiovirus, EMCV, und ein Apthovirus, das Maul- und Klauenseuchevirus (FMDV) teilen etwa 50 % identische IRES-Elemente. Das HCV-ähnliche Picornavirus-IRES weist den größten Unterschied in den IRES-Elementen von den anderen drei Klassen auf. Es gibt eine Vielzahl von Picornaviridae- Viren mit hochkonservierten HCV-ähnlichen IRES-Elementen, von denen einige noch identifiziert werden sollen. Es ist wichtig zu beachten, dass sich die HAV-IRES-Aktivität von den anderen drei Klassen durch ihre spezifische Anforderung an ein intaktes eIF4G unterscheidet. Andere Picornaviren kodieren für Proteine, die das eIF4G für eine verstärkte IRES-Aktivität spalten.
