Hierarchisches Clustering - Hierarchical clustering
| Teil einer Serie über |
|
Maschinelles Lernen und Data Mining |
|---|
 |
In Data Mining und Statistik ist hierarchisches Clustering (auch hierarchische Clusteranalyse oder HCA genannt ) eine Methode der Clusteranalyse, die versucht, eine Hierarchie von Clustern aufzubauen . Strategien für hierarchisches Clustering lassen sich im Allgemeinen in zwei Arten unterteilen:
- Agglomerativ : Dies ist ein " Bottom-up "-Ansatz: Jede Beobachtung beginnt in einem eigenen Cluster, und Clusterpaare werden zusammengeführt, wenn man in der Hierarchie aufsteigt.
- Divisiv : Dies ist ein " top-down "-Ansatz: Alle Beobachtungen beginnen in einem Cluster, und Aufteilungen werden rekursiv durchgeführt, wenn man sich in der Hierarchie nach unten bewegt.
Im Allgemeinen werden die Zusammenführungen und Aufteilungen gierig bestimmt . Die Ergebnisse der hierarchischen Clusterbildung werden üblicherweise in einem Dendrogramm dargestellt .
Der Standardalgorithmus für hierarchisches agglomeratives Clustering (HAC) hat eine Zeitkomplexität von und benötigt Speicher, was ihn selbst für mittlere Datensätze zu langsam macht. Für einige Spezialfälle sind jedoch optimale effiziente agglomerative Verfahren (von Komplexität ) bekannt: SLINK für Single-Linkage und CLINK für Complete -Linkage-Clustering . Mit einem Heap kann die Laufzeit des allgemeinen Falls auf , eine Verbesserung gegenüber der oben genannten Schranke von , reduziert werden , auf Kosten einer weiteren Erhöhung des Speicherbedarfs. In vielen Fällen ist der Speicher-Overhead dieses Ansatzes zu groß, um ihn praktisch nutzbar zu machen.
Mit Ausnahme des Sonderfalls Single-Linkage kann keiner der Algorithmen (außer der erschöpfenden Suche in ) garantiert die optimale Lösung finden.
Divisives Clustering mit einer erschöpfenden Suche ist , aber es ist üblich, schnellere Heuristiken zu verwenden, um Splits auszuwählen, wie z. B. k- means .
Clusterunähnlichkeit
Um zu entscheiden, welche Cluster kombiniert werden sollen (für agglomerative) oder wo ein Cluster aufgeteilt werden soll (für divisive), ist ein Maß für die Unähnlichkeit zwischen den Beobachtungssätzen erforderlich. Bei den meisten hierarchischen Clustering-Verfahren wird dies durch die Verwendung einer geeigneten Metrik (ein Maß für den Abstand zwischen Beobachtungspaaren) und eines Verknüpfungskriteriums erreicht, das die Unähnlichkeit von Mengen als Funktion der paarweisen Abstände von Beobachtungen in den Mengen angibt.
Metrisch
Die Wahl einer geeigneten Metrik beeinflusst die Form der Cluster, da einige Elemente unter einer Metrik relativ näher beieinander liegen können als unter einer anderen. In zwei Dimensionen ist beispielsweise unter der Manhattan-Distanzmetrik der Abstand zwischen dem Ursprung (0,0) und (0,5, 0,5) derselbe wie der Abstand zwischen dem Ursprung und (0, 1), während unter dem euklidischen Abstand metrisch ist die letztere strikt größer.
Einige häufig verwendete Metriken für hierarchisches Clustering sind:
Für Text oder andere nicht numerische Daten werden häufig Metriken wie die Hamming-Distanz oder die Levenshtein-Distanz verwendet.
Euklidische und Manhattan- Abstände sind die Spezialfälle der verallgemeinerten Minkowski-Distanz mit p = 1 (für Manhattan) und p = 2 (für euklidisch).
Es gibt mehrere andere Unähnlichkeitsmaße. Insbesondere korrelationsbasierte Distanzen – Pearson-, Eisen-Cosinus-, Spearman-, Kendall-Korrelationsdistanzen, die häufig für Genexpressionsdatenanalysen verwendet werden. Die korrelationsbasierte Distanz wird durch Subtraktion des Korrelationskoeffizienten von 1 definiert. Streng genommen können korrelationsbasierte Distanzen nicht als Metrik verwendet werden, die Quadratwurzel jedoch schon.
Eine Überprüfung der Clusteranalyse in der gesundheitspsychologischen Forschung ergab, dass das häufigste Distanzmaß in veröffentlichten Studien in diesem Forschungsbereich die euklidische Distanz oder die quadrierte euklidische Distanz ist.
Verknüpfungskriterien
Das Verknüpfungskriterium bestimmt den Abstand zwischen Beobachtungssätzen als Funktion der paarweisen Abstände zwischen den Beobachtungen.
Einige häufig verwendete Verknüpfungskriterien zwischen zwei Beobachtungssätzen A und B sind:
| Namen | Formel |
|---|---|
| Clustering mit maximaler oder vollständiger Verknüpfung | |
| Minimal- oder Single-Linkage-Clustering | |
| Ungewichtetes durchschnittliches Linkage-Clustering (oder UPGMA ) | |
| Gewichtetes durchschnittliches Linkage-Clustering (oder WPGMA ) | |
| Centroid Linkage Clustering oder UPGMC | wobei und die Schwerpunkte der Cluster s bzw. t sind. |
| Clustering mit minimaler Energie |
wobei d die gewählte Metrik ist. Weitere Verknüpfungskriterien sind:
- Die Summe aller Intra-Cluster-Varianzen.
- Die Zunahme der Varianz für den zusammenzuführenden Cluster ( Ward-Kriterium ).
- Die Wahrscheinlichkeit, dass Kandidatencluster aus derselben Verteilungsfunktion hervorgehen (V-Verknüpfung).
- Das Produkt von In-Grad und Out-Grad in einem k-nächsten-Nachbar-Graphen (Graphengradverknüpfung).
- Das Inkrement eines Cluster-Deskriptors (dh eine zum Messen der Qualität eines Clusters definierte Größe) nach dem Zusammenführen von zwei Clustern.
Diskussion
Hierarchisches Clustering hat den entscheidenden Vorteil, dass jedes gültige Distanzmaß verwendet werden kann. Tatsächlich sind die Beobachtungen selbst nicht erforderlich: Es wird lediglich eine Abstandsmatrix verwendet.
Beispiel für agglomeratives Clustering
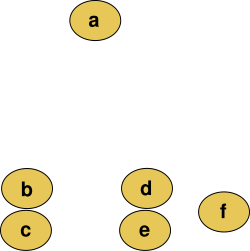
Angenommen, diese Daten sollen geclustert werden und die euklidische Distanz ist die Distanzmetrik .
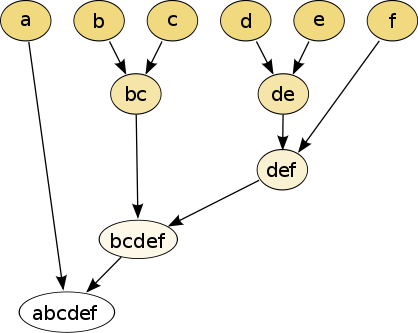
Das hierarchische Clustering- Dendrogramm wäre wie folgt :
Das Schneiden des Baums in einer bestimmten Höhe ergibt eine Partitionierungs-Clusterbildung mit einer ausgewählten Genauigkeit. In diesem Beispiel ergibt das Schneiden nach der zweiten Reihe (von oben) des Dendrogramms die Cluster {a} {bc} {de} {f}. Das Schneiden nach der dritten Reihe ergibt Cluster {a} {bc} {def}, was eine gröbere Clusterung mit einer kleineren Anzahl, aber größeren Clustern ist.
Diese Methode baut die Hierarchie aus den einzelnen Elementen auf, indem Cluster nach und nach zusammengeführt werden. In unserem Beispiel haben wir sechs Elemente {a} {b} {c} {d} {e} und {f}. Der erste Schritt besteht darin, zu bestimmen, welche Elemente in einem Cluster zusammengeführt werden sollen. Normalerweise möchten wir die beiden nächstgelegenen Elemente entsprechend der gewählten Entfernung nehmen.
Optional kann man in diesem Stadium auch eine Distanzmatrix konstruieren , wobei die Zahl in der i- ten Zeile j- ten Spalte der Abstand zwischen dem i- ten und j- ten Element ist. Wenn die Clusterbildung fortschreitet, werden dann Zeilen und Spalten zusammengeführt, während die Cluster zusammengeführt und die Abstände aktualisiert werden. Dies ist eine gängige Methode zur Implementierung dieser Art von Clustering und hat den Vorteil, dass Distanzen zwischen Clustern zwischengespeichert werden. Ein einfacher agglomerativer Clustering-Algorithmus wird auf der Single-Linkage-Clustering- Seite beschrieben; es kann leicht an verschiedene Gestängearten angepasst werden (siehe unten).
Angenommen, wir haben die beiden nächsten Elemente b und c verschmolzen , wir haben jetzt die folgenden Cluster { a }, { b , c }, { d }, { e } und { f } und wollen sie weiter verschmelzen. Dazu müssen wir den Abstand zwischen {a} und {bc} nehmen und damit den Abstand zwischen zwei Clustern definieren. Normalerweise ist der Abstand zwischen zwei Clustern und einer der folgenden:
- Der maximale Abstand zwischen den Elementen jedes Clusters (auch als Complete-Linkage-Clustering bezeichnet ):
- Der Mindestabstand zwischen den Elementen jedes Clusters (auch Single-Linkage-Clustering genannt ):
- Der mittlere Abstand zwischen den Elementen jedes Clusters (auch Average Linkage Clustering genannt, verwendet zB in UPGMA ):
- Die Summe aller Intra-Cluster-Varianzen.
- Die Zunahme der Varianz für den zusammenzuführenden Cluster ( Methode von Ward )
- Die Wahrscheinlichkeit, dass Kandidatencluster aus derselben Verteilungsfunktion hervorgehen (V-Verknüpfung).
Bei gebundenen Mindestabständen wird zufällig ein Paar ausgewählt, wodurch mehrere strukturell unterschiedliche Dendrogramme generiert werden können. Alternativ können alle gebundenen Paare gleichzeitig verbunden werden, wodurch ein eindeutiges Dendrogramm erzeugt wird.
Man kann immer dann entscheiden, das Clustering zu beenden, wenn eine genügend kleine Anzahl von Clustern vorhanden ist (Anzahlkriterium). Einige Verknüpfungen können auch garantieren, dass die Agglomeration in einem größeren Abstand zwischen den Clustern auftritt als die vorherige Agglomeration, und dann kann man die Clusterung beenden, wenn die Cluster zu weit voneinander entfernt sind, um zusammengeführt zu werden (Abstandskriterium). Dies ist jedoch zB bei der Schwerpunktverknüpfung nicht der Fall, wo die sogenannten Umkehrungen (Inversionen, Abweichungen von der Ultrametrie) auftreten können.
Trennendes Clustering
Das Grundprinzip des divisiven Clusterings wurde als DIANA-Algorithmus (DIvisive ANAlysis Clustering) veröffentlicht. Anfänglich befinden sich alle Daten im selben Cluster, und der größte Cluster wird geteilt, bis jedes Objekt getrennt ist. Da es Möglichkeiten gibt , jeden Cluster aufzuteilen, sind Heuristiken erforderlich. DIANA wählt das Objekt mit der maximalen durchschnittlichen Unähnlichkeit aus und verschiebt dann alle Objekte in dieses Cluster, die dem neuen Cluster ähnlicher sind als dem Rest.
Software
Open-Source-Implementierungen

- ALGLIB implementiert mehrere hierarchische Clustering-Algorithmen (Single-Link, Complete -Link, Ward) in C++ und C# mit O(n²) Speicher und O(n³) Laufzeit.
- ELKI umfasst mehrere hierarchische Clustering-Algorithmen, verschiedene Linkage-Strategien und umfasst auch die effizienten SLINK-, CLINK- und Anderberg-Algorithmen, flexible Clusterextraktion aus Dendrogrammen und verschiedene andere Clusteranalysealgorithmen .
- Octave , das GNU- Analogon zu MATLAB, implementiert hierarchisches Clustering in der Funktion "linkage".
- Orange , eine Data-Mining-Softwaresuite, umfasst hierarchisches Clustering mit interaktiver Dendrogramm-Visualisierung.
- R verfügt über viele Pakete, die Funktionen für hierarchisches Clustering bereitstellen.
- SciPy implementiert hierarchisches Clustering in Python, einschließlich des effizienten SLINK-Algorithmus.
- scikit-learn implementiert auch hierarchisches Clustering in Python.
- Weka beinhaltet hierarchische Clusteranalysen.
Kommerzielle Implementierungen
- MATLAB beinhaltet hierarchische Clusteranalyse.
- SAS beinhaltet die hierarchische Clusteranalyse in PROC CLUSTER.
- Mathematica enthält ein hierarchisches Clustering-Paket.
- NCSS beinhaltet hierarchische Clusteranalyse.
- SPSS umfasst eine hierarchische Clusteranalyse.
- Qlucore Omics Explorer beinhaltet eine hierarchische Clusteranalyse.
- Stata umfasst eine hierarchische Clusteranalyse.
- CrimeStat enthält einen hierarchischen Cluster-Algorithmus für den nächsten Nachbarn mit einer grafischen Ausgabe für ein geografisches Informationssystem.
Siehe auch
- Binäre Raumpartitionierung
- Begrenzende Volume-Hierarchie
- Braune Clusterbildung
- Kladistik
- Clusteranalyse
- Computergestützte Phylogenetik
- CURE-Datenclustering-Algorithmus
- Dasguptas Ziel
- Dendrogramm
- Bestimmung der Anzahl von Clustern in einem Datensatz
- Hierarchisches Clustering von Netzwerken
- Lokalitätsabhängiges Hashing
- Nächster Nachbar suchen
- Nearest-Neighbor-Chain-Algorithmus
- Numerische Taxonomie
- OPTIK-Algorithmus
- Statistische Distanz
- Persistente Homologie
Verweise
Weiterlesen
- Kaufmann, L.; Rousseeuw, PJ (1990). Finden von Gruppen in Daten: Eine Einführung in die Clusteranalyse (1 Hrsg.). New York: John Wiley. ISBN 0-471-87876-6.
- Hast, Trevor ; Tibshirani, Robert ; Friedman, Jerome (2009). "14.3.12 Hierarchisches Clustering" . Die Elemente des statistischen Lernens (2. Aufl.). New York: Springer. S. 520–8. ISBN 978-0-387-84857-0. Archiviert vom Original (PDF) am 10.11.2009 . Abgerufen 2009-10-20 .