Vertico räumlich modulierte Beleuchtung - Vertico spatially modulated illumination
Die räumlich modulierte Beleuchtung von Vertico ( Vertico-SMI ) ist das schnellste Lichtmikroskop für die 3D-Analyse kompletter Zellen im Nanometerbereich . Es basiert auf zwei 1996 entwickelten Technologien, SMI (räumlich modulierte Beleuchtung) und SPDM (spektrale Präzisionsdistanzmikroskopie). Die effektive optische Auflösung dieses optischen Nanoskops hat in 2D die Nähe von 5 nm und in 3D von 40 nm erreicht und die λ / 2-Auflösungsgrenze (etwa 200 nm für blaues Licht), die für die Standardmikroskopie unter Verwendung von Transmission oder Reflexion von natürlichem Licht gilt, weit überschritten (im Gegensatz zu strukturierter Beleuchtung oder Fluoreszenz ) gemäß der Abbe-Auflösungsgrenze Diese Grenze (auch als Rayleigh-Grenze bekannt ) wurde 1873 von Ernst Abbe festgelegt und regelt die erreichbare Auflösungsgrenze von Mikroskopen unter Verwendung herkömmlicher Techniken.
Das Vertico-SMI-Mikroskop wurde von einem Team unter der Leitung von Christoph Cremer , emeritiert an der Universität Heidelberg , entwickelt und basiert auf der Kombination von lichtoptischen Techniken der Lokalisationsmikroskopie (SPDM, spektrale Präzisionsdistanzmikroskopie ) und strukturierter Beleuchtung (SMI, räumlich modulierte Beleuchtung) ).
Seit März 2008 können mit dieser sogenannten SPDMphymod-Lokalisationsmikroskopie (physikalisch modifizierbare Fluorophore) viele Standard-Fluoreszenzfarbstoffe wie GFP- und Alexa-Fluoreszenzfarbstoffe verwendet werden, für die nur eine einzige Laserwellenlänge geeigneter Intensität für das Nanoimaging ausreicht.
Aufbau
SMI steht für eine spezielle Art der optischen Laserbeleuchtung ( räumlich modulierte Beleuchtung ) und Vertico spiegelt die vertikale Anordnung der Mikroskopachse wider, die die Analyse von festen Zellen, aber auch von lebenden Zellen mit einer optischen Auflösung unter 10 Nanometern (1 Nanometer = 1) ermöglicht nm = 1 × 10 –9 m).
Eine Besonderheit dieser Technologie im Vergleich zu Fokussiertechniken wie der 4Pi-Mikroskopie sind die Weitfeldaufnahmen, mit denen ganze Zellen im Nanomaßstab dargestellt werden können. Eine solche 3D-Belichtung einer ganzen Zelle mit einer typischen Objektgröße von 20 um × 20 um erfordert nur 2 Minuten. Weitfeldaufnahmen bedeuten, dass das gesamte Objekt gleichzeitig beleuchtet und erkannt wird.
Räumlich modulierte Beleuchtung
Die SMI-Mikroskopie ist ein lichtoptischer Prozess des sogenannten Point Spread Function Engineering. Hierbei handelt es sich um Prozesse, die die Punktstreufunktion (PSF) eines Mikroskops in geeigneter Weise modifizieren , um entweder die optische Auflösung zu erhöhen, die Genauigkeit der Abstandsmessungen von fluoreszierenden Objekten zu maximieren, die im Verhältnis zur Wellenlänge des Beleuchtungslichts klein sind , oder um andere Strukturparameter im Nanometerbereich extrahieren.
Das am Kirchhoff-Institut für Physik der Universität Heidelberg entwickelte SMI-Mikroskop erreicht dies folgendermaßen: Die Beleuchtungsstärke innerhalb des Objektbereichs ist im Gegensatz zu herkömmlichen Weitfeld-Fluoreszenzmikroskopen nicht gleichmäßig, sondern wird durch den Einsatz präzise räumlich moduliert von zwei gegenüberliegenden störenden Laserstrahlen entlang der Achse. Das Prinzip des räumlich modulierten Wellenfeldes wurde 1993 von Bailey et al. Der in der Heidelberg-Anwendung verwendete SMI-Mikroskopie-Ansatz bewegt das Objekt in hochpräzisen Schritten durch das Wellenfeld, oder das Wellenfeld selbst wird durch Phasenverschiebung relativ zum Objekt bewegt. Dies führt zu einer verbesserten axialen Größe und Abstandsauflösung.
SMI kann mit anderen Super-Resolution-Technologien kombiniert werden, beispielsweise mit 3D LIMON oder LSI- TIRF als Totalreflexionsinterferometer mit seitlicher Strukturbeleuchtung. Diese SMI-Technik ermöglichte es, lichtoptische Bilder von Autofluorophorverteilungen in den Schnitten aus menschlichem Augengewebe mit einer zuvor unerreichten optischen Auflösung aufzunehmen. Die Verwendung von drei verschiedenen Anregungswellenlängen (488, 568 und 647 nm) ermöglicht die Erfassung spektraler Informationen über das Autofluoreszenzsignal. Dies wurde für menschliches Augengewebe verwendet, das von AMD der Makuladegeneration betroffen ist .
SPDM: Lokalisationsmikroskopie
Eine einzelne, winzige Lichtquelle kann viel besser lokalisiert werden als die Auflösung eines Mikroskops: Obwohl das Licht einen verschwommenen Fleck erzeugt, können Computeralgorithmen verwendet werden, um die Mitte des verschwommenen Flecks unter Berücksichtigung der Punktstreufunktion genau zu berechnen des Mikroskops, die Rauscheigenschaften des Detektors und so weiter. Dieser Ansatz funktioniert jedoch nicht, wenn zu viele Quellen nahe beieinander liegen: Die Quellen verschwimmen alle miteinander.
SPDM (Spectral Precision Distance Microscopy) ist eine Familie von Techniken in der Fluoreszenzmikroskopie, die dieses Problem umgeht, indem jeweils nur wenige Quellen gemessen werden, sodass jede Quelle von den anderen "optisch isoliert" ist (dh durch mehr als die voneinander getrennt ist Auflösung des Mikroskops, typischerweise ~ 200-250 nm). Dann kann die obige Technik (Finden der Mitte jedes verschwommenen Flecks) verwendet werden.
Wenn die Moleküle eine Vielzahl unterschiedlicher Spektren (Absorptionsspektren und / oder Emissionsspektren) aufweisen, ist es möglich, Licht von jeweils nur wenigen Molekülen unter Verwendung der geeigneten Lichtquellen und Filter zu betrachten. Moleküle können auch auf subtilere Weise anhand der Fluoreszenzlebensdauer und anderer Techniken unterschieden werden.
Die mit SPDM erreichbare strukturelle Auflösung kann als kleinster messbarer Abstand zwischen zwei in ihrer räumlichen Position bestimmten punktförmigen Teilchen unterschiedlicher spektraler Eigenschaften ausgedrückt werden ("topologische Auflösung"). Modellierung hat in Bezug auf der Präzision der Lokalisierung, Teilchendichte etc. , die unter geeigneten Bedingungen dargestellt, die „topologische Auflösung“ entspricht eine „ Raumfrequenz “ , die im Sinne der klassischen Definition zu einer deutlich verbesserten optischen Auflösung entsprechen.
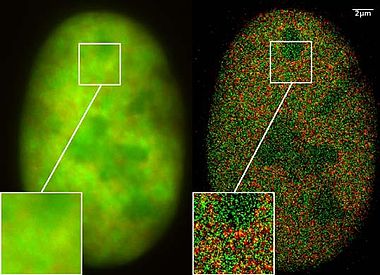
SPDM ist eine Lokalisierungsmikroskopie, die eine effektive optische Auflösung erzielt, die um ein Vielfaches besser ist als die herkömmliche optische Auflösung (ca. 200-250 nm), dargestellt durch die Halbwertsbreite des Hauptmaximums der effektiven Punktbildfunktion. Durch Anwendung geeigneter laseroptischer Präzisionsprozesse können Position und Abstände, die wesentlich kleiner als die halbe Breite der Punktstreufunktion (üblicherweise 200-250 nm) sind, mit Nanometergenauigkeit zwischen Zielen mit unterschiedlichen spektralen Signaturen gemessen werden. Ein wichtiges Anwendungsgebiet ist die Genomforschung (Untersuchung der funktionellen Organisation des Genoms ). Ein weiteres wichtiges Anwendungsgebiet ist die Erforschung der Struktur von Membranen.
Eine der wichtigsten Grundlagen der Lokalisationsmikroskopie im Allgemeinen ist die erste experimentelle Arbeit zur Lokalisierung fluoreszierender Objekte im Nanobereich (3D) im Jahr 1996 und ein theoretischer und experimenteller Beweis für eine Lokalisierungsgenauigkeit unter Verwendung von sichtbarem Licht im Bereich von 1 nm - die Basis für die Lokalisationsmikroskopie besser als 1/100 der Wellenlänge.
SPDMphymod: Standard-Fluoreszenzfarbstoffe im Blinkmodus wie GFP
Erst in den letzten zwei Jahren wurden Moleküle in nanoskopischen Untersuchungen verwendet, die dieselbe spektrale Lichtfrequenz (jedoch mit unterschiedlichen spektralen Signaturen basierend auf den Blitzeigenschaften) emittieren, aber mittels Licht ein- und ausgeschaltet werden können, wie es für die spektrale Präzision erforderlich ist Distanzmikroskopie. Durch die Kombination von vielen tausend Bildern derselben Zelle konnten mithilfe von laseroptischen Präzisionsmessungen Lokalisierungsbilder mit deutlich verbesserter optischer Auflösung aufgezeichnet werden. Die Anwendung dieser neuartigen Nanoskopieverfahren erschien bis vor kurzem sehr schwierig, da angenommen wurde, dass nur speziell hergestellte Moleküle unter Verwendung von Licht in geeigneter Weise ein- und ausgeschaltet werden können.
Im März 2008 entdeckte das Labor von Christoph Cremer , dass dies auch für viele Standard-Fluoreszenzfarbstoffe wie GFP , Alexa-Farbstoffe und Fluoresceinmoleküle möglich ist, sofern bestimmte photophysikalische Bedingungen vorliegen. Mit dieser sogenannten SPDMphymod-Technologie (physikalisch modifizierbare Fluorophore) reicht eine einzige Laserwellenlänge geeigneter Intensität für das Nanoimaging aus. Im Gegensatz dazu benötigen andere Lokalisierungsmikroskopien zwei Laserwellenlängen, wenn spezielle photoschaltbare / photoaktivierbare Fluoreszenzmoleküle verwendet werden.
Das GFP - Gen wurde in vielen prokaryotischen und eukaryotischen Zellen und der eingeführt und exprimiert Nobelpreis in Chemie 2008 wurde verliehen an Martin Chalfie , Osamu Shimomura , und Roger Y. Tsien für die Entdeckung und Entwicklung des grün fluoreszierenden Proteins. Die Erkenntnis, dass diese Standard-Fluoreszenzmoleküle verwendet werden können, erweitert die Anwendbarkeit der SPMD-Methode auf zahlreiche Forschungsbereiche in der Biophysik , Zellbiologie und Medizin .
Standard-Fluoreszenzfarbstoffe, die bereits erfolgreich mit der SPDMphymod-Technologie eingesetzt wurden: GFP, RFP, YFP, Alexa 488, Alexa 568, Alexa 647, Cy2, Cy3, Atto 488 und Fluorescein.
LIMON: 3D-Mikroskopie mit Superauflösung
LIMON (Light MicrOscopical Nanosizing Microscopy) wurde 2001 an der Universität Heidelberg erfunden und kombiniert Lokalisierungsmikroskopie und räumlich modulierte Beleuchtung mit der 3D-Mikroskopie mit hoher Auflösung.
Die 3D-Bilder mit Vertico-SMI werden durch die Kombination von SMI und SPDM ermöglicht, wobei zuerst der SMI- und dann der SPDM-Prozess angewendet werden. Der SMI-Prozess bestimmt das Zentrum der Partikel und ihre Ausbreitung in Richtung der Mikroskopachse. Während das Zentrum von Partikeln / Molekülen mit einer Genauigkeit von 1-2 nm bestimmt werden kann, kann die Ausbreitung um diesen Punkt bis zu einem axialen Durchmesser von ca. 30-40 nm.
Anschließend wird die laterale Position der einzelnen Partikel / Moleküle mit SPDM bestimmt, wobei eine Genauigkeit von wenigen Nanometern erreicht wird. Derzeit erreicht SPDM 16 Bilder / s mit einer effektiven Auflösung von 10 nm in 2D (Objektebene); Ungefähr 2000 solcher Bilder werden mit SMI-Daten (ca. 10 Sekunden Erfassungszeit) kombiniert, um ein dreidimensionales Bild mit höchster Auflösung (effektive optische 3D-Auflösung ca. 40-50 nm) zu erhalten. Mit einer schnelleren Kamera kann man noch höhere Raten erwarten (bis zu mehreren hundert Bildern / Sek., In Entwicklung). Mit geeigneten Farbstoffen sollten noch effektivere optische 3D-Auflösungen möglich sein
Durch die Kombination von SPDMphymod mit SMI (beide 1996 in Christoph Cremers Labor erfunden) wurde eine 3D-Zweifarbenrekonstruktion der räumlichen Anordnung von Her2 / neu- und Her3-Clustern erreicht. Die Positionen in allen drei Richtungen der Proteincluster konnten mit einer Genauigkeit von etwa 25 nm bestimmt werden.
Einsatz der hochauflösenden Mikroskopie in der Industrie
Trotz des Einsatzes in biomedizinischen Labors könnten Super-Resolution-Technologien als wichtige Werkzeuge in der pharmazeutischen Forschung dienen. Sie könnten besonders hilfreich bei der Identifizierung und Bewertung von Zielen sein. Beispielsweise sind biomolekulare Maschinen (BMM) hochkomplexe Nanostrukturen, die aus mehreren großen Molekülen bestehen und für die Grundfunktionen in den Körperzellen verantwortlich sind. Abhängig von ihrem Funktionsstatus haben sie eine definierte 3D-Struktur. Beispiele für biomolekulare Maschinen sind Nukleosomen, die es der DNA, einem zwei Meter langen Träger genetischer Information, ermöglichen, die Körperzellen nur in einem Raum von einigen Millionstel Millimetern Durchmesser zu falten. Daher kann die DNA als Informations- und Kontrollzentrum dienen.
Durch die Verwendung von LIMON 3D in Kombination mit der Markierung des LIMON-Komplexes ist es erstmals möglich, versteckte Proteine oder Nukleinsäuren eines 3D-Molekülkomplexes der sogenannten biomolekularen Maschinen sichtbar zu machen, ohne den Komplex zu zerstören. Bisher bestand das Problem in den meisten Fällen darin, dass der Komplex für eine detaillierte Analyse der einzelnen darin enthaltenen Makromoleküle zerstört werden musste. Alternativ wurden virtuelle Computersimulationsmodelle oder teure Kernspinresonanzmethoden verwendet, um die dreidimensionale Struktur solcher Komplexe zu visualisieren.
Literatur
Externe Links
- GFP Superresolution
- Publikationsliste optische Nanoskopie
- Pressemitteilung der Universität Heidelberg